Diferencia entre revisiones de «Transcripción»
| Línea 1: | Línea 1: | ||
{{Definición | {{Definición | ||
|nombre= Transcripción | |nombre= Transcripción | ||
| − | |imagen= | + | |imagen= Transcrpcion.jpg |
|tamaño= | |tamaño= | ||
|concepto= Proceso en el que se sintetiza ARN a partir de una plantilla de ADN, permitiendo la transferencia de información genética para la síntesis de proteínas (traducción). | |concepto= Proceso en el que se sintetiza ARN a partir de una plantilla de ADN, permitiendo la transferencia de información genética para la síntesis de proteínas (traducción). | ||
}} | }} | ||
| − | La ''transcripción'' es un proceso celular esencial en el que se sintetiza una molécula de [[ARN]] complementaria a una secuencia de [[ADN]], facilitando la expresión génica. El ARN resultante (llamado ''transcrito'') sigue las reglas de complementariedad de bases. | + | La '''transcripción''' es un proceso celular esencial en el que se sintetiza una molécula de [[ARN]] complementaria a una secuencia de [[ADN]], facilitando la expresión génica. El ARN resultante (llamado ''transcrito'') sigue las reglas de complementariedad de bases. |
| − | + | == Diferencias entre dominios biológicos == | |
* ''Bacterias'': | * ''Bacterias'': | ||
| − | + | ** Transcripción y [[traducción]] ocurren ''acopladas'' en el citoplasma (sin núcleo). | |
| − | + | ** Velocidad: ~40 nucleótidos/segundo (Dulin ''et al.'', 2015). | |
* ''Eucariotas'': | * ''Eucariotas'': | ||
| − | + | ** La transcripción ocurre en el [[núcleo celular|núcleo]]. | |
| − | + | ** La traducción ocurre en el citoplasma. | |
| − | + | ** Requiere procesamiento del ARN (ej. ''splicing''). | |
{| class="wikitable" style="margin: 1em auto;" | {| class="wikitable" style="margin: 1em auto;" | ||
| Línea 34: | Línea 34: | ||
=== Iniciación === | === Iniciación === | ||
* ''Promotores'': | * ''Promotores'': | ||
| − | + | ** Bacterias: ''Caja de Pribnow'' (TATAAT) en posición -10 (Pribnow, 1975). | |
| − | + | ** Eucariotas: ''Caja TATA'' (TATAAA) en posición -25. | |
* ''Factores de transcripción'': | * ''Factores de transcripción'': | ||
| − | + | ** [[ARN polimerasa]] se une al promotor (Alberts ''et al.'', 2017). | |
| − | + | ** En eucariotas: Complejo de iniciación con factores TFII. | |
=== Elongación === | === Elongación === | ||
| Línea 47: | Línea 47: | ||
=== Terminación === | === Terminación === | ||
* ''Bacterias'': | * ''Bacterias'': | ||
| − | + | ** ''Independiente de Rho'': Horquilla GC + cola de uracilos. | |
| − | + | ** ''Dependiente de Rho'': Hexámero ρ reconoce secuencias "rut". | |
* ''Eucariotas'': | * ''Eucariotas'': | ||
| − | + | ** Señal de poliadenilación (AAUAAA) (Lodish ''et al.'', 2016). | |
| − | + | ** Corte y adición de cola poli-A. | |
== Regulación == | == Regulación == | ||
Revisión del 20:10 31 mar 2025
| ||||||
La transcripción es un proceso celular esencial en el que se sintetiza una molécula de ARN complementaria a una secuencia de ADN, facilitando la expresión génica. El ARN resultante (llamado transcrito) sigue las reglas de complementariedad de bases.
Sumario
Diferencias entre dominios biológicos
- Bacterias:
- Transcripción y traducción ocurren acopladas en el citoplasma (sin núcleo).
- Velocidad: ~40 nucleótidos/segundo (Dulin et al., 2015).
- Eucariotas:
- La transcripción ocurre en el núcleo.
- La traducción ocurre en el citoplasma.
- Requiere procesamiento del ARN (ej. splicing).
| Base en ADN | Base en ARN |
|---|---|
| A | U |
| T | A |
| C | G |
| G | C |
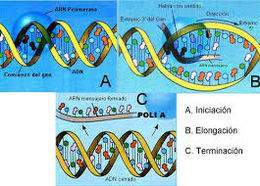
Etapas de la transcripción
Iniciación
- Promotores:
- Bacterias: Caja de Pribnow (TATAAT) en posición -10 (Pribnow, 1975).
- Eucariotas: Caja TATA (TATAAA) en posición -25.
- Factores de transcripción:
- ARN polimerasa se une al promotor (Alberts et al., 2017).
- En eucariotas: Complejo de iniciación con factores TFII.
Elongación
- Formación de la burbuja de transcripción (~14 pb desenrolladas).
- Síntesis en dirección 5' → 3'.
- En eucariotas: Modificaciones co-transcripcionales (5' cap, poli-A).
Terminación
- Bacterias:
- Independiente de Rho: Horquilla GC + cola de uracilos.
- Dependiente de Rho: Hexámero ρ reconoce secuencias "rut".
- Eucariotas:
- Señal de poliadenilación (AAUAAA) (Lodish et al., 2016).
- Corte y adición de cola poli-A.
Regulación
- Elementos cis: Secuencias reguladoras (enhancers/silencers).
- Factores trans: Proteínas que se unen a elementos reguladores (Allis & Jenuwein, 2016).
- Modificaciones epigenéticas (metilación, acetilación).
Técnicas de estudio
- Pulso-caza: Uso de nucleótidos marcados (³H-uridina).
- Run-on transcription: Mide transcripción activa.
- RNA-seq: Secuenciación masiva de transcritos (Wang et al., 2009).
Fuentes
- Alberts, B., Johnson, A., Lewis, J., Morgan, D., Raff, M., Roberts, K. & Walter, P. (2017). Molecular biology of the cell (6ª ed.). Garland Science. https://doi.org/10.1201/9781315735368
- Lodish, H., Berk, A., Kaiser, C. A., Krieger, M., Bretscher, A., Ploegh, H., Amon, A. & Martin, K. C. (2016). Molecular cell biology (8ª ed.). W.H. Freeman. ISBN 978-1464183393.
- National Center for Biotechnology Information. (2023). Eukaryotic transcription control. https://www.ncbi.nlm.nih.gov/books/NBK9904/ (Consultado el 31 de marzo de 2025).
- Nature Education. (2014). Transcription: DNA to RNA. Scitable. https://www.nature.com/scitable/topicpage/transcription-dna-to-rna-393/ (Consultado el 31 de marzo de 2025).
- Allis, C. D. & Jenuwein, T. (2016). The molecular hallmarks of epigenetic control. Nature Reviews Genetics, 17(8), 487-500. https://doi.org/10.1038/nrg.2016.59
- Wang, Z., Gerstein, M. & Snyder, M. (2009). RNA-Seq: a revolutionary tool for transcriptomics. Nature Reviews Genetics, 10(1), 57-63. https://doi.org/10.1038/nrg2484
- Pribnow, D. (1975). Nucleotide sequence of an RNA polymerase binding site at an early T7 promoter. Proceedings of the National Academy of Sciences, 72(3), 784-788. https://doi.org/10.1073/pnas.72.3.784
- Dulin, D., Bauer, D. L. V., Malinen, A. M., Bakermans, J. J. W., Kaller, M., Morichaud, Z., Petushkov, I., Depken, M., Brodolin, K. & Kulbachinskiy, A. (2015). Pause sequences facilitate entry into long-lived paused states by reducing RNA polymerase transcription rates. Nature Communications, 6, 10157. https://doi.org/10.1038/ncomms10157