ADN mitocondrial
| ||||
El ADN Mitocondrial (ADNmt) o genoma mitocondrial es un material genético de las mitocondrias, los elementos de la célula que generan energía para la misma. Se trata de un material genético circular cerrado de doble cadena que se localiza en el interior de las mitocondrias celulares.
Sumario
Generalidades
La mitocondria es un orgánulo de probable origen endosimbióntico que se ha adaptado a su nicho intracelular: para aumentar su tasa de replicación y asegurar la transmisión a las células hijas después de cada división mitótica, el genoma de las mitocondrias de mamíferos se ha ido reduciendo de tamaño hasta alcanzar las 16.569 kb en el caso del genoma mitocondrial humano. Las mitocondrias son las verdaderas centrales térmicas de nuestro organismo ya que en ellas tiene lugar la fosforilación oxidativa (OXPHOS), es decir, la respiración celular acoplada a la producción de energía en forma de ATP. El funcionamiento del sistema OXPHOS tiene, además, importancia médica por la generación de especies reactivas de O2 (Reactive Oxygen Species, ROS) y por la regulación de la muerte celular programada o apoptosis. Las proteínas incluidas en el OXPHOS se localizan dentro de la membrana mitocondrial interna, e incluyen:
- Componentes de la cadena transportadora de electrones (Cadena respiratoria mitocondrial, CRM);
- ATPasa de membrana;
- Translocador de nucleótidos de Adenina (ANT).
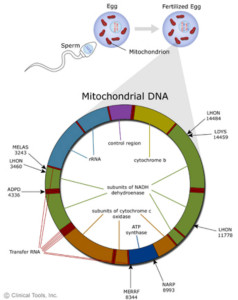
El ADNmt humano es una [[molécula++ circular de 16.569 pares de bases. El número de moléculas de ADNmt por célula varía entre unos pocos cientos en los espermatozoides a unas 200.000 copias en el oocito, pero en la mayor parte de los tejidos el rango está comprendido entre unas 1.000 y 10.000 copias por célula, con 2 - 10 moléculas de ADN por mitocondria. Este genoma contiene información para 37 genes: • Genes que codifican las 2 subunidades 12S y 16S del ARNr (ARN ribosomal) de la matriz mitocondrial. • Los genes para los 22 ARNt (ARN transferente), requeridos para la síntesis de proteínas mitocondriales en la misma matriz mitocondrial. • Genes que codifican 13 polipéptidos que forman parte de los complejos multienzimáticos del sistema OXPHOS. En concreto, en el genoma mitocondrial se codifican 7 subunidades del Complejo I, 1 subunidad del Complejo III, 3 subunidades del Complejo IV, y 2 subunidades de la ATPasa (Complejo V). Es importante no perder de vista que el resto de las subunidades polipeptídicas de estos complejos, así como el Complejo II completo, están codificados en el genoma nuclear, de manera que no todas las enfermedades mitocondriales están necesariamente causadas por alteraciones en el ADN mitocondrial. La característica estructural más sorprendente del ADNmt es que los genes se encuentran situados uno a continuación del otro, sin apenas intrones ni regiones no codificantes entre los genes. Al contrario que el genoma nuclear, en el que las regiones no codificantes son mayoritarias, el ADN mitocondrial sólo posee un 3% de secuencias no codificantes. Veintiocho de los genes mitocondriales (2 ARNr, 14 ARNt y 12 polipéptidos) se encuentran en una de las cadenas (cadena H ó pesada), mientras que los 9 genes restantes (1 polipéptido y 8 ARNt) están en la cadena complementaria (cadena L ó ligera). La única zona del ADNmt que no codifica ningún gen es la región del bucle de desplazamiento (bucle-D), localizada alrededor del origen de replicación de la cadena H. Esta región contiene también los promotores de la transcripción y los elementos reguladores de la expresión génica. Otra de las peculiaridades de la organización genética del ADNmt es que los genes de los ARNt se distribuyen entre los genes de los ARNr y los codificantes de proteínas; esta disposición tiene consecuencias muy importantes para el procesamiento del ARN. Para la replicación del ADNmt hacen falta dos orígenes diferentes, uno para cada cadena (OH y OL). Ambos orígenes de replicación están muy separados, haciendo que el proceso sea unidireccional y asimétrico. La síntesis del ADN se inicia en OH y es realizada por una polimerasa específica de la mitocondria, la DNApol ?, que alarga un ARN iniciador fruto del procesamiento de un transcrito primario que se sintetiza a partir del promotor L. La replicación continúa de modo unidireccional hasta alcanzar OL, momento en el cual comienza la síntesis de la segunda cadena del ADN, alargando también un pequeño iniciador de ARN. En la transcripción del ADNmt intervienen una polimerasa de ARN, al menos un factor de transcripción implicado en la iniciación (mtTFA), y uno de terminación (mTERF). Las dos cadenas del ADNmt se transcriben completamente a partir de tres puntos de iniciación diferentes, dos para la cadena pesada (H1 y H2) y uno para la cadena ligera (L), originando tres moléculas policistrónicas que se procesan posteriormente por cortes endonucleolíticos precisos en los extremos 5´ y 3´ de las secuencias de los ARNt, para dar lugar a los ARNr, ARNt y ARNm maduros. De esta forma los ARNt, situados entre los genes de los ARNr y ARNm, actúan como señales de reconocimiento para los enzimas de procesamiento. En particular, la cadena H se transcribe mediante dos unidades de transcripción solapadas en la región de los ARNr: la primera de estas unidades comienza delante del gen para el ARNtPhe (lugar de iniciación H1), termina en el extremo 3´ del gen para el ARNr 16S y es responsable de la síntesis de los ARNr 12S y 16S, del ARNtPhe y del ARNtVal. El factor de terminación (mTERF) se une a una secuencia situada en el gen del ARNtLeu y provoca la terminación de esta unidad. La segunda unidad de transcripción comienza cerca del extremo 5´ del gen del ARNr 12S (lugar de iniciación H2) y transcribe la casi totalidad de la cadena pesada; el procesamiento de este ARN policistrónico origina los ARNm de 12 péptidos y los otros 12 ARNt codificados en esta cadena. La transcripción de la cadena ligera comienza cerca del extremo 5´ del ARN 7S (en el bucle-D) y da lugar al iniciador de la replicación de la cadena pesada, 8 ARNt y 1 péptido (ND6). La síntesis de las proteínas mitocondriales tiene lugar en ribosomas específicos de la mitocondria, cuyos componentes están codificados en el ADNmt (ARNr 12S y 16S) y en el genoma nuclear (84 proteínas ribosomales). En este sistema de traducción se sintetizan las trece proteínas codificadas en el ADNmt utilizando un código genético que difiere ligeramente del código genético universal. Así, UGA codifica el aminoácido triptófano (Trp) en vez de ser un codón de terminación, y los codones AUA y AUU se utilizan también como codones de iniciación. La biogénesis de la mitocondria depende de la expresión coordinada de los genomas mitocondrial y nuclear, pero hasta ahora se conoce muy poco acerca de los mecanismos que regulan la interacción de ambos sistemas genéticos. La expresión del ADNmt parece estar regulada por el factor de iniciación de la transcripción mtTFA, codificado en el genoma nuclear. Este factor podría ser el responsable tanto de los niveles de ARN como del número de copias de ADNmt, ya que la replicación depende de la síntesis de un iniciador de ARN a partir del promotor de la cadena ligera. La regulación de la relación entre los ARNr y los ARNm mitocondriales se realiza fundamentalmente mediante la selección del lugar de iniciación de la transcripción de la cadena pesada, que a su vez está relacionada con el factor mtTERF (que causa terminación de la transcripción después de la síntesis de los ARNr) y con el procesamiento de los ARN primarios. Asimismo, la actividad transcripcional puede estar regulada por estímulos hormonales, especialmente por hormonas tiroideas que actúan tanto de un modo indirecto (por activación de genes nucleares) como directamente sobre el propio ADNmt.
Análisis del ADN Mitocondrial para estudios antropológicos y evolutivos
El ADN mitocondrial fue descubierto por Margit Nass y Sylvan Nass, utilizando microscopia electrónica y un marcador sensitivo al ADN mitocondrial. Este ADN mitocondrial es un pequeño trozo de ADN que se encuentra en las estructuras fuera del núcleo, recibiendo estas estructuras el nombre de mitocondrias. Estas son en realidad antiguas bacterias que entraron en las células tempranas cuando aún eran células vivas individuales (hace aproximadamente unos tres millones de años), las cuales han formado una relación simbiótica en las que las mitocondrias viven a modo de bacterias dentro de nuestras células. Una vez que estas bacterias tienen su propio sistema de información, que es el ADN, convierten esta información en las estructuras en forma de ARN y luego en proteínas. Pero como comenta el genetista molecular Douglas Wallace, ‹‹Lo importante es que la mitocondria y el ADN mitocondrial no se transmiten a lo largo de los cromosomas que se encuentran en la mayoría de los genes de las células, pero se pasan por el óvulo de la madre en la fertilización. Esto es debido a que el óvulo tiene alrededor de 200.000 mitocondrias, mientras que los espermatozoides poseen sólo unos pocos, y los que consiguen entrar en el óvulo son destruidos. Por lo tanto, la mitocondria y su ADN se heredan exclusivamente de la madre.››
El ADNmt contiene información genética sólo de la madre, mientras que el nuclear contiene información de ambos padres.
Este estudio de ADN se puede llegar a realizar gracias a la gran cantidad de mitocondrias que contiene una célula (un centenar), e incluso dentro de cada mitocondria coexisten entre 1000 y 10000 copias de ADN mitocondrial. Por lo tanto, y debido a que en este tipo de estudios el ADN suele ser muy escaso y estar muy degradado (por ejemplo, cuando se realizan estudios de ADN antiguo), la gran cantidad de ADN mitocondrial en una célula hace que su recuperación sea más eficiente que la que encontramos en el ADN nuclear o autosómico. Otro uso de este tipo de genoma, y además cada vez más empleado, es la identificación de cadáveres en los cuales debido a su gran estado de descomposición, no puede ser descubierta su identidad. En muchas ocasiones se utilizan también otros marcadores monoparentales (es decir, transmitidos de padres/madres a hijos) pero también a través del cromosoma ‹‹Y›› (transmitido por parte paterna). El problema viene con las limitaciones que en ocasiones presentan este tipo de muestras debido tanto a la conservación como a las propias contaminaciones que la misma pueda sufrir. A pesar de la gran importancia que tiene el uso de los marcadores monoparentales en medicina forense, lo cierto es que con los últimos años su uso para conocer el ADN de restos esqueletizados de gran antigüedad han supuesto una verdadera revolución para el estudio de la antropología física y de la arqueología. Por lo tanto, el estudio de las poblaciones de la antigüedad puede llegar a ser investigado con una cierta efectividad en el caso de que tengamos tanto un estado de conservación básico de ciertos restos óseos, así como la posibilidad de obtener una muestra amplia, pues es necesario y recomendable su comparación con distintos individuos de la misma procedencia arqueológica. Así también, la actual posibilidad de recuperar ADN de especies animales extintas ha permitido obtener evidencias directas de algunas características genéticas de las poblaciones pasadas. Además, las dificultades derivadas de las técnicas empleadas con este tipo de ADN se han conseguido superar gracias al desarrollo de la reacción en cadena de la polimerasa (PCR), una técnica que permite ser usada con muestras biológicas que poseen muy poco ADN o cuando se encuentre muy degradado, como es el caso normal de los restos arqueológicos. A pesar de la gran importancia de esta técnica para la obtención de una datación más concreta, debemos tener cuidado a la hora de utilizarla. Esto se debe a que la compresión filogenética más antigua debe tratarse con sumo cuidado, pues no puede de ser observada nunca desde una perspectiva actual, ya que hay que tener en cuenta diversos factores que no se pueden obtener a través de estudios genéticos como los cambios culturales, migraciones, u otras evidencias de tipo arqueológico o antropológico. A pesar de todo, poco a poco estos problemas se van superando y podemos decir de forma general que el avance en los estudios de las relaciones filogenéticas en muchas ocasiones proporciona una mayor información y más exacta que los estudios de tipo osteométricos (es decir, a través de la información que proporcionan las medidas de los restos óseos). Dentro de los trabajos más destacados donde se han utilizado marcadores monoparentales nos encontramos el realizado con la secuenciación de un fragmento del genoma mitocondrial de un espécimen de neandertal, hallado en 1845 en Dusseldorf. Este estudio indicaba que la secuencia difiere tanto del hombre como del chimpancé, apoyando por lo tanto el origen africano reciente del hombre moderno (en torno a los 100.000 años). Actualmente también se trabaja con otra metodología para el estudio del ADNmt basado en la sencillez metodológica en comparación con la secuenciación, lo que abre nuevas posibilidades a los estudios de tipo poblacional. Este consiste en establecer lo que se conoce como haplogrupos, los cuales nos pueden mostrar las distintas variables geográficas de la especie humana (caucasoides, africanos, asiáticos…), lo que ofrece una mayor facilidad para inferir relaciones evolutivas entre las poblaciones. En cuanto a las muestras usadas para los estudios del genoma mitocondrial, los investigadores coinciden en que deben de usarse muestras lo menos contaminadas posibles. Por lo tanto, y a pesar de que hasta hace relativamente poco tiempo estas muestras se obtenían del tejido blando (especialmente de tejido momificado y tejido de especies animales preservados en museos), a día de hoy los tejidos óseos y dentales pueden ofrecer una mayor información genética. Un ejemplo del uso del ADNmt lo encontramos en el yacimiento paleolítico del Pirulejo (Córdoba), donde sus autores nos explican en profundidad todos los métodos empleados para la obtención de distintas muestras de ADNmt de este yacimiento. En esta excavación se realizó de una manera exhaustiva la comparación de muestras del yacimiento con muestras ya conocidas a priori, como fue la introducción en el análisis de muestras de dos aborígenes de la Tierra del Fuego. Esto le proporcionó mayor veracidad al proceso de análisis y sus resultados. Además, gracias a la creación de los haplogrupos anteriormente citados, se pudo concluir que los restos pertenecían a un haplogrupo de regiones del Próximo Oriente, más concretamente de Macedonia, lo que resultaría totalmente coherente siguiendo la idea de esta zona como de paso obligado durante las expansiones humanas. Estos resultados, con sólo estudios antropométricos, no se hubieran podido conocer. A modo de conclusión, en este artículo se ha podido resumir la importancia que tiene actualmente el uso de los marcadores monoparentales, especialmente del ADNmt por su mayor amplitud. Por lo tanto, se ha demostrado de una forma muy eficaz la gran utilidad que este tipo de análisis tienen para establecer coincidencias genéticas con muestras esqueletizadas muy antiguas, e incluso (y de ahí su gran importancia), con muestras que apenas poseen ADN útil para su investigación. A pesar de todo los problemas que los investigadores se pueden encontrar, está claro que el uso del ADNmt nos proporciona una información hasta hace poco tiempo inimaginable y nos permite establecer linajes genéticos que nos acercan aún más al misterio que supone el estudio de la evolución humana.
Tasa de mutación del ADN mitocondrial
El ADN mitocondrial codifica 13 proteínas involucradas en la producción de energía celular y procesos de fosforilación oxidativa. Por lo tanto, el entorno que rodea la mitocondria y el ADN mitocondrial está expuesto al daño oxidativo producido por los radicales libres generados en ese metabolismo. Si a esto se le añade el hecho de que el material genético de las mitocondrias no está protegido por histonas como lo está el ADN nuclear, y que los mecanismos de reparación de daños el ADN son poco eficientes en las mitocondrias, obtenemos como resultado que la tasa de mutación aumenta hasta ser 10 veces mayor que la del genoma nuclear.
Herencia
Tradicionalmente se ha considerado que el ADN mitocondrial humano se hereda sólo por vía materna. Según esta concepción, cuando un espermatozoide fecunda un óvulo penetra el núcleo y su cola junto con sus mitocondrias son destruidos en el óvulo materno. Por lo tanto, en el desarrollo del cigoto sólo intervendrían las mitocondrias contenidas en el óvulo. Sin embargo, se ha demostrado que las mitocondrias del espermatozoide pueden ingresar al óvulo. Según algunos autores el ADN mitocondrial del padre puede perdurar en algunos tejidos, como los músculos. Según otros, no llega a heredarse al ser marcado por ubiquitinación y degradado.
Usos
El ADN mitocondrial puede ser usado para identificar individuos junto con otra evidencia. También es usado por laboratorios forenses para caracterizar viejas muestras de esqueleto humano. Distinto que el ADN nuclear, el ADN mitocondrial no sirve para identificar individuos sin ambigüedad, pero si para detectar parentescos entre grupos de individuos; es usado entonces para comparaciones entre personas desaparecidas y restos no identificados y sus familiares.
ADNmt para determinar parentescos
El ADN mitocondrial humano tiene características únicas que lo hacen muy apropiado para estudios microevolutivos: la herencia del genoma mitocondrial se realiza exclusivamente por la vía materna, sin recombinarse; hay un fragmento en este genoma de 400pb (pares de bases) altamente polimórfico, y posee una alta frecuencia de mutaciones (5 a 10 veces mayor que el ADN nuclear). Este ADN se puede extraer de muestras de cualquier tejido, incluso de la sangre y del tejido óseo. Gracias a su presencia en el hueso se puede obtener el genoma de individuos ya muertos desde hace muchos años. El análisis de la secuencia genómica se usa para estudiar las relaciones filogenéticas, no sólo en humanos sino, también en muchos otros organismos. Por este motivo se utiliza para determinar variabilidad en poblaciones naturales (para ver si hay o no endogamia), información útil para la conservación de especies en peligro de extinción. Otras aplicaciones Hay estudios de investigación que utilizan genes mitocondriales que pueden ocasionar algún tipo de enfermedad. Algunos investigadores defienden que es posible que la tendencia a la obesidad se herede por genes mitocondriales de vía materna. Este descubrimiento supone una vía de actuación contra este problema si se consiguiera regular el ADN mitocondrial con ciertos fármacos. El genoma mitocondrial posee infinidad de ventajas para estudiar relaciones evolutivas: Debido a su menor tamaño, el estudio del ADNmt es más fácil que el del ADN nuclear; además se puede extraer en grandes cantidades, porque cada célula tiene varias mitocondrias. El ADNmt evoluciona más rápido y no se recombina, pasando intacto entre generaciones salvo por las mutaciones; facilitando la identificación de las relaciones entre organismos muy parecidos.
Fuentes
- IZAGUIRRE, N.; DURAN, N.L; DE LA RUA, C., “Genética y Arqueología: Análisis molecular de ADN procedente de restos esqueléticos”. Revista Munibe, nº50, San Sebastián, 1998.
- DNA Interactive. www.dnai.org
- VALERA,T.A., AÍNSUA, R.L., FARIÑA, J., ”El ADN mitocondrial y las relaciones filogenéticas de los últimos estadios del género homo”, Revista Real Academia Galega de Ciencias, Volumen XXVIII, Pág. 103-118, 2009.
- CRESPILLO MÁRQUEZ, M.; BAÑÓN GONZÁLEZ, R.; VALVERDE DE VILLAREAL, J.L., “Aprendizaje y reflexiones de la identificación de cadáveres mediante marcadores genéticos monoparentales (ADNmt, cromosoma Y). A propósito de un caso”, Revista Española de Medicina Legal, nº 37 (1), 17-21, 2011.
- ADN Antiguo, Universidad Complutense de Madrid. http://pendientedemigracion.ucm.es/info/genforen/tecnicas1.htm
- TURBÓN, D.; CORTÉS, M.; FERNÁNDEZ, E.; PRATS, E., “Análisis del ADN mitocondrial de dos muestras del yacimiento paleolítico del Pirulejo”, Antiqvitas, nº20, pág. (193-198), M. H. M.: Priego de Córdoba, 2008.
- GRIFFITHS, A.J.F.; MILLER, J. H.; SUZUKI, D.T.; LEWONTIN, R.C.; GELBART, W. M., Genética, Séptima edición, McGran- Hill- Interamericana.