ADN
| ||||||||
El ADN o ácido desoxirribonucleico ―del inglés deoxyribonucleic acid (DNA)― es un tipo de ácido nucleico, una macromolécula que forma parte de todas las células.
Contiene la información genética usada en el desarrollo y el funcionamiento de los organismos vivos conocidos y de algunos virus, siendo el responsable de su transmisión hereditaria.
ADN
Desde el punto de vista químico, el ADN es un polímero de nucleótidos, es decir, un polinucleótido. Un polímero es un compuesto formado por muchas unidades simples conectadas entre sí, como si fuera un largo tren formado por vagones. En el ADN, cada vagón es un nucleótido, y cada nucleótido, a su vez, está formado por un azúcar (la desoxirribosa), una base nitrogenada (que puede ser
un grupo fosfato que actúa como enganche de cada vagón con el siguiente.
Lo que distingue a un vagón (nucleótido) de otro es, entonces, la base nitrogenada, y por ello la secuencia del ADN se especifica nombrando sólo la secuencia de sus bases. La disposición secuencial de estas cuatro bases a lo largo de la cadena (el ordenamiento de los cuatro tipos de vagones a lo largo de todo el tren) es la que codifica la información genética: por ejemplo, una secuencia de ADN puede ser ATGCTAGATCGC...
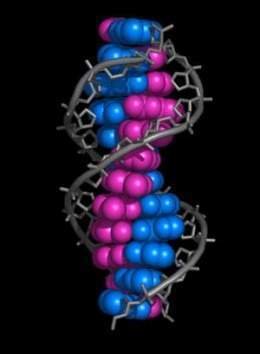
En los organismos vivos, el ADN se presenta como una doble cadena de nucleótidos, en la que las dos hebras están unidas entre sí por unas conexiones denominadas puentes de hidrógeno. Para que la información que contiene el ADN pueda ser utilizada por la maquinaria celular, debe copiarse en primer lugar en unos trenes de nucleótidos, más cortos y con unas unidades diferentes, llamados ARN. Las moléculas de ARN se copian exactamente del ADN mediante un proceso denominado transcripción. Una vez procesadas en el núcleo celular, las moléculas de ARN pueden salir al citoplasma para su utilización posterior.
La información contenida en el ARN se interpreta usando el Código genético, que especifica la secuencia de los Aminoácidos de las proteínas, según una correspondencia de un triplete de nucleótidos (Codón) para cada aminoácido. Esto es, la información genética (esencialmente: qué proteínas se van a producir en cada momento del ciclo de vida de una célula) se halla codificada en las secuencias de nucleótidos del ADN y debe traducirse para poder ser empleada. Tal traducción se realiza empleando el código genético a modo de diccionario. El diccionario "secuencia de nucleótido-secuencia de aminoácidos" permite el ensamblado de largas cadenas de aminoácidos (las proteínas) en el citoplasma de la célula.
Por ejemplo, en el caso de la secuencia de ADN indicada antes (ATGCTAGATCGC...), la ARN polimerasa utilizaría como molde la cadena complementaria de dicha secuencia de ADN (que sería TAC-GAT-CTA-GCG-...) para transcribir una molécula de ARNm que se leería AUG-CUA-GAU-CGC-... ; el ARNm resultante, utilizando el código genético, se traduciría como la secuencia de aminoácidos Metionina-Leucina-Ácido aspártico-Arginina-...
Las secuencias de ADN que constituyen la unidad fundamental, física y funcional de la herencia se denominan Genes. Cada gen contiene una parte que se transcribe a ARN y otra que se encarga de definir cuándo y dónde deben expresarse. La información contenida en los genes (genética) se emplea para generar ARN y Proteínas, que son los componentes básicos de las células, los "ladrillos" que se utilizan para la construcción de los orgánulos celulares, entre otras funciones.
Dentro de las células, el ADN está organizado en estructuras llamadas Cromosomas que, durante el Ciclo celular, se duplican antes de que la célula se divida. Los organismos eucariotas (por ejemplo, Animales, Plantas, y hongos) almacenan la inmensa mayoría de su ADN dentro del Núcleo celular y una mínima parte en los elementos celulares llamados Mitocondrias y Cloroplastos, y los Centros Organizadores de Microtúbulos o Centríolos, en caso de tenerlos; los organismos procariotas (Bacterias y Arqueas) lo almacenan en el Citoplasma de la célula, y, por último, los Virus ADN lo hacen en el interior de la cápsida de naturaleza proteica.
Existen multitud de proteínas, como por ejemplo las Histonas y los factores de transcripción, que se unen al ADN dotándolo de una estructura tridimensional determinada y regulando su expresión. Los factores de transcripción reconocen secuencias reguladoras del ADN y especifican la pauta de transcripción de los genes. El material genético completo de una dotación cromosómica se denomina Genoma y, con pequeñas variaciones, es característico de cada especie.
Historia
El ADN fue aislado por vez primera durante el invierno de 1869 por el médico suizo Friedrich Miescher mientras trabajaba en la Universidad de Tubinga. Miescher realizaba experimentos acerca de la composición química del Pus de vendas quirúrgicas desechadas cuando notó un precipitado de una sustancia desconocida que caracterizó químicamente más tarde. Se necesitaron casi 70 años de investigación para poder identificar los componentes y la estructura de los ácidos nucleicos. En 1919 Phoebus Levene identificó que un nucleótido está formado por una base, un Azúcar y un Fosfato. Sin embargo, Levene pensaba que la cadena era corta y que las bases se repetían en un orden fijo. En 1937 William Astbury produjo el primer patrón de Difracción de rayos X que mostraba que el ADN tenía una estructura regular.
La función biológica del ADN comenzó a dilucidarse en 1928, con una serie básica de experimentos de la genética moderna realizados por Frederick Griffith, quien estaba trabajando con cepas "lisas" (S) o "rugosas" (R) de la bacteria Pneumococcus (causante de la neumonía), según la presencia (S) o no (R) de una cápsula azucarada que es la que confiere virulencia (véase también Experimento de Griffith).
La inyección de neumococos S vivos en ratones produce la muerte de éstos, y Griffith observó que si inyectaba ratones con neumococos R vivos o con neumococos S muertos por calor, los ratones no morían. Sin embargo, si inyectaba a la vez neumococos R vivos y neumococos S muertos, los ratones morían, y en su sangre se podían aislar neumococos S vivos.
Como las bacterias muertas no pudieron haberse multiplicado dentro del ratón, Griffith razonó que debía producirse algún tipo de cambio o transformación de un tipo bacteriano a otro por medio de una transferencia de alguna sustancia activa, que denominó "principio transformante". Esta sustancia proporcionaba la capacidad a los neumococos R de producir una cápsula azucarada y transformarse así en virulentas. En los siguientes 15 años, estos experimentos iniciales fueron duplicados mezclando distintos tipos de cepas bacterianas muertas por el calor con otras vivas, tanto en ratones (in vivo) como en tubos de ensayo (in vitro).
La búsqueda del «factor transformante» que era capaz de hacer virulentas a cepas que inicialmente no lo eran continuó hasta 1944, año en el cual Oswald Avery, Colin MacLeod y Maclyn McCarty realizaron un experimento hoy clásico. Estos investigadores extrajeron la fracción activa (el factor transformante), y mediante análisis químicos, enzimáticos y serológicos, observaron que no contenía proteínas, ni lípidos no ligados, ni polisacáridos activos, sino que estaba constituido principalmente por "una forma viscosa de ácido desoxirribonucleico altamente polimerizado", es decir, ADN.
El ADN extraído de las cepas bacterianas S muertas por el calor lo mezclaron "in vitro" con cepas R vivas: el resultado fue que se formaron colonias bacterianas S, por lo que se concluyó inequívocamente que el factor o principio transformante era el ADN.
En cuanto a la caracterización química de la molécula, en 1940 Chargaff realizó algunos experimentos que le sirvieron para establecer las proporciones de las bases nitrogenadas en el ADN. Descubrió que las proporciones de purinas eran idénticas a las de pirimidinas; la "equimolecularidad" de las bases ([A]=[T], [G]=[C]) y que la cantidad de G+C en una determinada molécula de ADN no siempre es igual a la cantidad de A+T y puede variar desde el 36% al 70% del contenido total. Con esta información y junto con los datos de Difracción de rayos X proporcionados por Rosalind Franklin; James Watson y Francis Crick propusieron en 1953 el modelo de la doble hélice de ADN para representar la estructura tridimensional del polímero.
En 1962, después de la muerte de Franklin, los científicos Watson, Crick y Wilkins recibieron conjuntamente el Premio Nobel en Fisiología o Medicina.The Nobel Prize in Physiology or Medicine 1962 Nobelprize .org (Revisado el 22 de diciembre de 06). Sin embargo, el debate continúa sobre quién debería recibir crédito por el descubrimiento.
Propiedades físicas y químicas
El ADN es un largo Polímero formado por unidades repetitivas, los Nucleótidos. Una doble cadena de ADN mide de 22 a 26 Angstroms (2,2 a 2,6 Nanómetros) de ancho, y una unidad (un nucleótido) mide 3,3 Å (0,33 nm) de largo.
En los Organismos vivos, el ADN no suele existir como una molécula individual, sino como una pareja de moléculas estrechamente asociadas. Las dos cadenas de ADN se enroscan sobre sí mismas formando una especie de escalera de caracol, denominada Doble hélice. El modelo de estructura en doble hélice fue propuesto en 1953 por James Watson y Francis Crick (el artículo Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid fue publicado el 25 de abril de 1953 en Nature).Watson JD; Crick FHC. A structure for Deoxyribose Nucleic Acid. Nature 171(4356):737-738.. (April, 1953) Texto Completo
El éxito de éste modelo radicaba en su consistencia con las propiedades físicas y químicas del ADN. El estudio mostraba además que la complementariedad de bases podía ser relevante en su replicación, y también la importancia de la secuencia de bases como portador de información genética.Cada unidad que se repite, el nucleótido, contiene un segmento de la estructura de soporte (azúcar + fosfato), que mantiene la cadena unida, y una Base, que interacciona con la otra cadena de ADN en la hélice.
En general, una base ligada a un azúcar se denomina Nucleósido y una base ligada a un azúcar y a uno o más grupos fosfatos recibe el nombre de Nucleótido. Cuando muchos nucleótidos se encuentran unidos, como ocurre en el ADN, el polímero resultante se denomina Polinucleótido.Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN), consultado el 3 enero 2006
Composición del ADN
Estructura de soporte La estructura de soporte de una hebra de ADN está formada por unidades alternas de grupos Fosfato y azúcar.El azúcar en el ADN es una pentosa, concretamente, la Desoxirribosa.
- Su Fórmula química es H3PO4. Cada Nucleótido puede contener uno (monofosfato: AMP), dos (difosfato: ADP) o tres (trifosfato: ATP) grupos de ácido fosfórico, aunque como monómeros constituyentes de los Ácidos nucleicos sólo aparecen en forma de nucleósidos monofosfato.
- Es un Monosacárido de 5 Átomos de Carbono (una Pentosa) derivado de la Ribosa, que forma parte de la estructura de nucleótidos del ADN. Su fórmula es C5H10O4. Una de las principales diferencias entre el ADN y el ARN es el azúcar, pues en el ARN la 2-Desoxirribosa del ADN es reemplazada por una Pentosa alternativa, la Ribosa.
- Las moléculas de azúcar se unen entre sí a través de grupos fosfato, que forman enlaces fosfodiéster entre los átomos de carbono tercero (3′, «tres prima») y quinto (5′, «cinco prima») de dos anillos adyacentes de azúcar. La formación de enlaces asimétricos implica que cada hebra de ADN tiene una dirección.
En una Doble hélice, la dirección de los Nucleótidos en una hebra (3′ → 5′) es opuesta a la dirección en la otra hebra (5′ → 3′). Esta organización de las hebras de ADN se denomina antiparalela; son cadenas paralelas, pero con direcciones opuestas. De la misma manera, los extremos asimétricos de las hebras de ADN se denominan extremo 5′ («cinco prima») y extremo 3′ («tres prima») respectivamente.
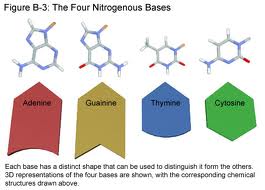
- Las cuatro bases nitrogenadas mayoritarias que se encuentran en el ADN son la Adenina (abreviado A), Citosina (C), Guanina (G) y Timina (T). Cada una de estas cuatro bases está unida al armazón de azúcar-fosfato a través del azúcar para formar el nucleótido completo (base-azúcar-fosfato).
Las bases son compuestos heterocíclicos y aromáticos con dos o más Átomos de Nitrógeno, y, dentro de las bases mayoritarias, se clasifican en dos grupos: las bases púricas o purinas (adenina y guanina), derivadas de la Purina y formadas por dos anillos unidos entre sí, y las bases pirimidínicas o pirimidinas (citosina y timina), derivadas de la Pirimidina y con un solo anillo.
En los ácidos nucleicos existe una quinta base pirimidínica, denominada Uracilo (U), que normalmente ocupa el lugar de la timina en el ARN y difiere de ésta en que carece de un grupo metilo en su anillo. El uracilo no se encuentra habitualmente en el ADN, sólo aparece raramente como un producto residual de la degradación de la citosina por procesos de desaminación oxidativa.
-
- En el Código genético se representa con la letra T. Es un derivado pirimidínico con un Grupo oxo en las posiciones 2 y 4, y un grupo metil en la posición 5. Forma el Nucleósido Timidina (siempre desoxitimidina ya que sólo aparece en el ADN) y el Nucleótido Timidilato o timidina monofosfato (dTMP).
En el ADN, la timina siempre se empareja con la adenina de la cadena complementaria mediante 2 puentes de hidrógeno, T=A. Su fórmula química es C5H6N2O2 y su nomenclatura 2, 4-dioxo, 5-metilpirimidina.
-
- En el código genético se representa con la letra C. Es un derivado pirimidínico, con un Grupo amino en posición 4 y un grupo oxo en posición 2. Forma el Nucleósido Citidina (desoxicitidina en el ADN) y el Nucleótido Citidilato o (desoxi)citidina monofosfato (dCMP en el ADN, CMP en el ARN).
La citosina siempre se empareja en el ADN con la guanina de la cadena complementaria mediante un triple enlace, C≡G. Su fórmula química es C4H5N3O y su nomenclatura 2-oxo, 4 aminopirimidina. Su Masa molecular es de 111,10 unidades de masa atómica. La citosina fue descubierta en 1894 cuando fue aislada en tejido del Timo de carnero.
-
- En el código genético se representa con la letra A. Es un derivado de la purina con un grupo amino en la posición 6. Forma el nucleósido Adenosina (desoxiadenosina en el ADN) y el nucleótido Adenilato o (desoxi)adenosina monofosfato (dAMP, AMP). En el ADN siempre se empareja con la timina de la cadena complementaria mediante 2 puentes de hidrógeno, A=T. Su fórmula química es C5H5N5 y su nomenclatura 6-aminopurina. La adenina, junto con la timina, fue descubierta en 1885 por el médico alemán Albrecht Kossel.
- En el código genético se representa con la letra G. Es un derivado púrico con un grupo oxo en la posición 6 y un grupo amino en la posición 2. Forma el nucleósido (desoxi)Guanosina y el nucleótido Guanilato o (desoxi)guanosina monofosfato (dGMP, GMP). La guanina siempre se empareja en el ADN con la citosina de la cadena complementaria mediante tres enlaces de hidrógeno, G≡C. Su fórmula química es C5H5N5O y su nomenclatura 6-oxo, 2-aminopurina.
También existen otras bases nitrogenadas (las llamadas bases nitrogenadas minoritarias), derivadas de forma natural o sintética de alguna otra base mayoritaria. Lo son por ejemplo la Hipoxantina, relativamente abundante en el TRNA, o la Cafeína, ambas derivadas de la adenina; otras, como el Aciclovir, derivadas de la guanina, son análogos sintéticos usados en terapia antiviral; otras, como una de las derivadas del uracilo, son antitumorales.
Las bases nitrogenadas tienen una serie de características que les confieren unas propiedades determinadas. Una característica importante es su carácter aromático, consecuencia de la presencia en el anillo de dobles enlaces en posición conjugada. Ello les confiere la capacidad de absorber luz en la zona ultravioleta del espectro en torno a los 260 Nm, lo cual puede ser aprovechado para determinar el coeficiente de extinción del ADN y hallar la concentración existente de los ácidos nucleicos.
Otra de sus características es que presentan Tautomería o Isomería de grupos funcionales debido a que un átomo de Hidrógeno unido a otro átomo puede migrar a una posición vecina; en las bases nitrogenadas se dan dos tipos de tautomerías: tautomería lactama-lactima, donde el hidrógeno migra del nitrógeno al Oxígeno del grupo oxo (forma lactama) y viceversa (forma lactima), y tautomería imina-amina primaria, donde el hidrógeno puede estar formando el grupo amina (forma amina primaria) o migrar al nitrógeno adyacente (forma imina). La adenina sólo puede presentar tautomería amina imina, la timina y el uracilo muestran tautomería doble lactama-lactima, y la guanina y citosina pueden presentar ambas. Por otro lado, y aunque se trate de moléculas Apolares, las bases nitrogenadas presentan suficiente carácter polar como para establecer Puentes de hidrógeno, ya que tienen átomos muy Electronegativos (nitrógeno y oxígeno) presentando carga parcial negativa, y átomos de hidrógeno con carga parcial positiva, de manera que se forman dipolos que permiten que se formen estos enlaces débiles.
Se estima que el Genoma humano Haploide tiene alrededor de 3.000 millones de pares de bases. Para indicar el tamaño de las moléculas de ADN se indica el número de pares de bases, y como derivados hay dos unidades de medida muy utilizadas, la Kilobase (kb), que equivale a 1.000 pares de bases, y la Megabase (Mb), que equivale a un millón de pares de bases.
Apareamiento de bases
La dóble hélice de ADN se mantiene estable mediante la formación de puentes de hidrógeno entre las bases asociadas a cada una de las dos hebras. Para la formación de un Enlace de hidrógeno una de las bases debe presentar un "donador" de hidrógenos con un átomo de hidrógeno con carga parcial positiva (-NH2 o -NH) y la otra base debe presentar un grupo "aceptor" de hidrógenos con un átomo cargado electronegativamente (C=O o N). Los puentes de hidrógeno son uniones más débiles que los típicos enlaces químicos covalentes, como los que conectan los átomos en cada hebra de ADN, pero más fuertes que interacciones hidrófobas individuales, enlaces de Van der Waals, etc. Como los puentes de hidrógeno no son enlaces covalentes, pueden romperse y formarse de nuevo de forma relativamente sencilla. Por esta razón, las dos hebras de la doble hélice pueden separarse como una cremallera, bien por fuerza mecánica o por alta Temperatura.
Cada tipo de base en una hebra forma un enlace únicamente con un tipo de base en la otra hebra, lo que se denomina "complementariedad de las bases". Según esto, las purinas forman enlaces con las pirimidinas, de forma que A se enlaza sólo con T, y C sólo con G. La organización de dos nucleótidos apareados a lo largo de la doble hélice se denomina apareamiento de bases. Este emparejamiento corresponde a la observación ya realizada por Erwin Chargaff (1905-2002),Erwin Chargaff Papers que mostró que la cantidad de adenina era muy similar a la cantidad de timina, y que la cantidad de citosina era igual a la cantidad de guanina en el ADN.
Como resultado de esta complementariedad, toda la información contenida en la secuencia de doble hebra de la hélice de ADN está duplicada en cada hebra, lo cual es fundamental durante el proceso de replicación del ADN. En efecto, esta interacción reversible y específica entre pares de bases complementarias es crítica para todas las funciones del ADN en los organismos vivos.
Como se ha indicado anteriormente, los dos tipos de pares de bases forman un número diferente de enlaces de hidrógeno: A=T forman dos puentes de hidrógeno, y C≡G forman tres puentes de hidrógeno (ver imágenes). El par de bases GC es por tanto más fuerte que el par de bases AT. Como consecuencia, tanto el porcentaje de pares de bases GC como la longitud total de la doble hélice de ADN determinan la fuerza de la asociación entre las dos hebras de ADN.
Las dobles hélices largas de ADN con alto contenido en GC tienen hebras que interaccionan más fuertemente que las dobles hélices cortas con alto contenido en AT. Por esta razón, las zonas de la doble hélice de ADN que necesitan separarse fácilmente tienden a tener un alto contenido en AT, como por ejemplo la secuencia TATAAT de la Caja de Pribnow de algunos promotores.
En el laboratorio, la fuerza de esta interacción puede medirse buscando la temperatura requerida para romper los puentes de hidrógeno, la temperatura de fusión (también denominado valor Tm, del inglés melting temperature).
Cuando todas las pares de bases en una doble hélice se funden, las hebras se separan en solución en dos hebras completamente independientes. Estas moléculas de ADN de hebra simple no tienen una única forma común, sino que algunas conformaciones son más estables que otras.
Otros tipos de pares de bases
Existen diferentes tipos de pares de bases que se pueden formar según cómo se forman los puentes de hidrógeno. Los que encontramos en la doble hélice de ADN son los llamados pares de Bases Watson-Crick, pero también existen otros posibles pares de bases, como los denominados Hoogsteen y Wobble u oscilante, que pueden aparecer en circunstancias particulares. Además, para cada tipo existe a su vez el mismo par reverso, es decir, el que se da si se gira la base pirimidínica 180º sobre su eje.
- Watson-Crick (pares de bases de la doble hélice): los grupos de la base púrica que intervienen en el enlace de hidrógeno son los que corresponden a las posiciones 1 y 6 (N aceptor y -NH2 donador si la purina es una A) y los grupos de la base pirimidínica los que se encuentran en las posiciones 3 y 4 (-NH donador y C=O aceptor si la pirimidina es una T). En el par de bases Watson-Crick reverso participarían los grupos de las posiciones 2 y 3 de la base pirimidínica (ver imágenes).
- Hoogsteen: en este caso cambian los grupos de la base púrica, que ofrece una cara diferente (posiciones 6 y 7) y que forman enlaces con los grupos de las pirimidinas de las posiciones 3 y 4 (como en Watson-Crick). También puede haber Hoogsteen reversos. Con este tipo de enlace pueden unirse A=U (Hoogsteen y Hoogsteen reverso) y A=C (Hoogsteen reverso).
- Wobble u oscilante: este tipo de enlace permite que se unan guanina y citosina con un doble enlace (G=T). La base púrica (G) forma enlace con los grupos de las posiciones 1 y 6 (como en Watson-Crick) y la pirimidina (T) con los grupos de las posiciones 2 y 3. Este tipo de enlace no funcionaría con A=C, ya que quedarían enfrentados los 2 aceptores y los 2 donadores, y sólo se podría dar en el caso inverso. Encontramos pares de bases de tipo oscilante en el ARN, durante el apareamiento de Codón y Anticodón. Con este tipo de enlace pueden unirse G=U (oscilante y oscilante reverso) y A=C (oscilante reverso).
En total, en su forma tautomérica mayoritaria, existen 28 posibles pares de bases nitrogenadas: 10 posibles pares de bases purina-pirimidina (2 pares Watson-Crick y 2 Watson Crick reverso, 1 par Hoogsteen y 2 pares Hoogsteen reverso, 1 par oscilante y 2 pares oscilante reverso), 7 pares homo purina-purina (A=A, G=G), 4 pares A=G y 7 pares pirimidina-pirimidina. Esto sin contar con los pares de bases que pueden formarse si también tenemos en cuenta las otras formas tautoméricas minoritarias de las bases nitrogenadas; éstos, además, pueden ser responsables de mutaciones puntuales por sustitución de tipo transición.
Estructura
El ADN es una Molécula bicatenaria, es decir, está formada por dos cadenas dispuestas de forma antiparalela y con las bases nitrogenadas enfrentadas. En su estructura tridimensional, se distinguen distintos niveles:Hib, J. & De Robertis, E. D. P. 1998. Fundamentos De Biología Celular Y Molecular. El Ateneo, 3ª edición, 416 páginas. ISBN 950-02-0372-3. ISBN 978-950-02-0372-2De Robertis, E.D.P. 1998. Biología Celular Y Molecular. El Ateneo, 617 páginas. ISBN 950-02-0364-2. ISBN 978-950-02-0364-7
- Estructura primaria:
- Secuencia de nucleótidos encadenados. Es en estas cadenas donde se encuentra la información genética, y dado que el esqueleto es el mismo para todos, la diferencia de la información radica en la distinta secuencia de bases nitrogenadas. Esta secuencia presenta un código, que determina una información u otra, según el orden de las bases.
- Estructura secundaria:
- Es una estructura en doble hélice. Permite explicar el almacenamiento de la información genética y el mecanismo de duplicación del ADN. Fue postulada por Watson y Crick, basándose en la difracción de rayos X que habían realizado Franklin y Wilkins, y en la equivalencia de bases de Chargaff, según la cual, la suma de adeninas más guaninas es igual a la suma de timinas más citosinas.
- Es una cadena doble, dextrógira o levógira, según el tipo de ADN. Ambas cadenas son complementarias, pues la adenina y la guanina de una cadena se unen, respectivamente, a la timina y la citosina de la otra. Ambas cadenas son antiparalelas, pues el extremo 3´ de una se enfrenta al extremo 5´ de la homóloga.
- Existen tres modelos de ADN. El ADN de tipo B es el más abundante y es el descubierto por Watson y Crick.
- Estructura terciaria:
- Se refiere a cómo se almacena el ADN en un espacio reducido, para formar los Cromosomas. Varía según se trate de organismos Procariotas o Eucariotas:
- En Procariotas el ADN se pliega como una súper-hélice, generalmente en forma circular y asociada a una pequeña cantidad de proteínas. Lo mismo ocurre en Orgánulos celulares como las Mitocondrias y en los Cloroplastos.
- En Eucariotas, dado que la cantidad de ADN de cada Cromosoma es muy grande, el empaquetamiento ha de ser más complejo y compacto; para ello se necesita la presencia de proteínas, como las Histonas y otras proteínas de naturaleza no histónica (en los Espermatozoides estas proteínas son las Protaminas).
Estructuras en doble hélice
El ADN existe en muchas conformaciones. Sin embargo, en organismos vivos sólo se han observado las conformaciones ADN-A, ADN-B y ADN-Z. La conformación que adopta el ADN depende de su secuencia, la cantidad y dirección de superenrollamiento que presenta, la presencia de modificaciones químicas en las bases y las condiciones de la solución, tales como la concentración de Iones de Metales y Poliaminas.De las tres conformaciones, la forma "B" es la más común en las condiciones existentes en las células. Las dos dobles hélices alternativas del ADN difieren en su geometría y dimensiones.
La forma "A" es una espiral que gira hacia la derecha, más amplia que la "B", con una hendidura menor superficial y más amplia, y una hendidura mayor más estrecha y profunda. La forma "A" ocurre en condiciones no fisiológicas en formas deshidratadas de ADN, mientras que en la célula puede producirse en apareamientos híbridos de hebras ADN-ARN, además de en complejos enzima-ADN.
Los segmentos de ADN en los que las bases han sido modificadas por Metilación pueden sufrir cambios conformacionales mayores y adoptar la forma "Z". En este caso, las hebras giran alrededor del eje de la hélice en una espiral que gira a mano izquierda, lo opuesto a la forma "B" más frecuente. Estas estructuras poco frecuentes pueden ser reconocidas por proteínas específicas que se unen a ADN-Z y posiblemente estén implicadas en la regulación de la transcripción.
Estructuras en cuádruplex
En los extremos de los cromosomas lineales existen regiones especializadas de ADN denominadas Telómeros. La función principal de estas regiones es permitir a la célula replicar los extremos cromosómicos utilizando la enzima Telomerasa, puesto que las enzimas que replican el resto del ADN no pueden copiar los extremos 3' de los cromosomas. Estas terminaciones cromosómicas especializadas también protegen los extremos del ADN, y previenen que los sistemas de Reparación del ADN en la célula los procesen como ADN dañado que debe ser corregido.
En las células humanas, los telómeros son largas zonas de ADN de hebra sencilla que contienen algunos miles de repeticiones de una única secuencia TTAGGG. Estas secuencias ricas en guanina pueden estabilizar los extremos cromosómicos mediante la formación de estructuras de juegos apilados de unidades de cuatro bases, en lugar de los pares de bases encontrados normalmente en otras estructuras de ADN. En este caso, cuatro bases guanina forman unidades con superficie plana que se apilan una sobre otra, para formar una estructura cuádruplex-G estable. Estas estructuras se estabilizan formando puentes de hidrógeno entre los extremos de las bases y la quelatación de un metal iónico en el centro de cada unidad de cuatro bases. También se pueden formar otras estructuras, con el juego central de cuatro bases procedente, o bien de una hebra sencilla plegada alrededor de las bases, o bien de varias hebras paralelas diferentes, de forma que cada una contribuye una base a la estructura central.
Además de estas estructuras apiladas, los Telómeros también forman largas estructuras en lazo, denominadas lazos teloméricos o lazos-T (T-loops en inglés). En este caso, las hebras simples de ADN se enroscan sobre sí mismas en un amplio círculo estabilizado por proteínas que se unen a telómeros.En el extremo del lazo-T, el ADN telomérico de hebra sencilla se sujeta a una región de ADN de doble hebra porque la hebra de ADN telomérico altera la doble hélice y se aparea a una de las dos hebras. Esta estructura de triple hebra se denomina lazo de desplazamiento o lazo-D (D-loop).
Hendiduras mayor y menor
La Doble hélice es una espiral Dextrógira, esto es, cada una de las cadenas de Nucleótidos gira a derechas; esto puede verificarse si nos fijamos, yendo de abajo a arriba, en la dirección que siguen los segmentos de las hebras que quedan en primer plano. Si las dos hebras giran a derechas se dice que la doble hélice es dextrógira, y si giran a izquierdas, levógira (esta forma puede aparecer en hélices alternativas debido a cambios conformacionales en el ADN). Pero en la conformación más común que adopta el ADN, la doble hélice es dextrógira, girando cada par de bases respecto al anterior unos 36º.
Cuando las dos hebras de ADN se enrollan una sobre la otra (sea a derechas o a izquierdas), se forman huecos o hendiduras entre una hebra y la otra, dejando expuestos los laterales de las Bases nitrogenadas del interior (ver la animación). En la conformación más común que adopta el ADN aparecen, como consecuencia de los ángulos formados entre los azúcares de ambas cadenas de cada par de bases nitrogenadas, dos tipos de hendiduras alrededor de la superfície de la doble hélice: una de ellas, la hendidura o surco mayor, que mide 22 Å (2,2 Nm) de ancho, y la otra, la hendidura o surco menor, que mide 12 Å (1,2 nm) de ancho.
Cada vuelta de hélice, que es cuando ésta ha realizado un giro de 360º o lo que es lo mismo, de principio de hendidura mayor a final de hendidura menor, medirá por tanto 34 Å, y en cada una de esas vueltas hay unos 10,5 pb. left||300px|thumb|Hendiduras mayor y menor de la doble hélice. La anchura de la hendidura mayor implica que los extremos de las bases son más accesibles en ésta, de forma que la cantidad de grupos químicos expuestos también es mayor lo cual facilita la diferenciación entre los pares de bases A-T, T-A, C-G, G-C.
Como consecuencia de ello, también se verá facilitado el reconocimiento de secuencias de ADN por parte de diferentes Proteínas sin la necesidad de abrir la doble hélice. Así, proteínas como los factores de transcripción que pueden unirse a secuencias específicas, frecuentemente contactan con los laterales de las bases expuestos en la hendidura mayor.Por el contrario, los grupos químicos que quedan expuestos en la hendidura menor son similares, de forma que el reconocimiento de los pares de bases es más difícil; por ello se dice que la hendidura mayor contiene más información que la hendidura menor.
Sentido y antisentido
Una secuencia de ADN se denomina "sentido" (en inglés, sense) si su secuencia es la misma que la secuencia de un ARN mensajero que se traduce en una proteína. La secuencia de la hebra de ADN complementaria se denomina "antisentido" (antisense). En ambas hebras de ADN de la doble hélice pueden existir tanto secuencias sentido, que codifican ARNm, como antisentido, que no lo codifican, es decir, las secuencias que codifican ARNm no están todas presentes en una sola de las hebras, sino repartidas entre las dos hebras. Tanto en Procariotas como en Eucariotas se producen ARNs con secuencias antisentido, pero la función de esos ARNs no está completamente clara.
Se ha propuesto que los ARNs antisentido están implicados en la regulación de la Expresión génica mediante apareamiento ARN-ARN: los ARNs Antisentido se aparearían con los ARNm complementarios, bloqueando de esta forma su traducción.
En unas pocas secuencias de ADN en procariotas y eucariotas (este hecho es más frecuente en Plásmidos y Virus), la distinción entre hebras sentido y antisentido es más difusa, debido a que presentan genes superpuestos.En estos casos, algunas secuencias de ADN tienen una función doble, codificando una proteína cuando se lee a lo largo de una hebra, y una segunda proteína cuando se lee en la dirección contraria a lo largo de la otra hebra. En Bacterias, esta superposición puede estar involucrada en la regulación de la Transcripción del gen,mientras que en virus los genes superpuestos aumentan la cantidad de información que puede codificarse en sus diminutos genomas.
Superenrollamiento
El ADN puede retorcerse como una cuerda en un proceso que se denomina superenrollamiento del ADN («supercoiling», en inglés). Cuando el ADN está en un estado "relajado", una hebra normalmente gira alrededor del eje de la doble hélice una vez cada 10,4 pares de bases, pero si el ADN está retorcido las hebras pueden estar unidas más estrechamente o más relajadamente.
Si el ADN está retorcido en la dirección de la hélice, se dice que el superenrollamiento es positivo, y las bases se mantienen juntas de forma más estrecha. Si el ADN se retuerce en la dirección opuesta, el superenrollamiento se llama negativo, y las bases se alejan. En la naturaleza, la mayor parte del ADN tiene un ligero superenrollamiento negativo que es producido por Enzimas denominadas Topoisomerasas. Estas enzimas también son necesarias para liberar las fuerzas de torsión introducidas en las hebras de ADN durante procesos como la Transcripción y la Replicación.
Modificaciones de bases
La expresión de los genes está influenciada por la forma en la que el ADN está empaquetado en cromosomas, en una estructura denominada Cromatina. Las modificaciones de bases pueden estar implicadas en el empaquetamiento del ADN: las regiones que presentan una expresión génica baja o nula normalmente contienen niveles altos de Metilación de las bases Citosina. Por ejemplo, la metilación de citosina produce 5-metil-citosina, que es importante para la inactivación del Cromosoma X. El nivel medio de metilación varía entre organismos: el gusano Caenorhabditis elegans carece de metilación de citosina, mientras que los Vertebrados presentan un nivel alto - hasta 1% de su ADN contiene 5-metil-citosina.A pesar de la importancia de la 5-metil-citosina, ésta puede desAminarse para generar una base timina. Las citosinas metiladas son por tanto particularmente sensibles a mutaciones.Otras modificaciones de bases incluyen la metilación de Adenina en Bacterias y la Glicosilación de Uracilo para producir la "base-J" en Kinetoplastos.
Daño del ADN
El ADN puede resultar dañado por muchos tipos de Mutágenos, que cambian la secuencia del ADN: agentes alquilantes, además de Radiación electromagnética de alta energía, como luz Ultravioleta y Rayos X.
El tipo de daño producido en el ADN depende del tipo de mutágeno. Por ejemplo, la luz UV puede dañar al ADN produciendo dímeros de Timina, que se forman por ligamiento cruzado entre bases pirimidínicas. Por otro lado, oxidantes tales como radicales libres o el Peróxido de hidrógeno producen múltiples daños, incluyendo modificaciones de bases, sobre todo guanina, y roturas de doble hebra (double-strand breaks).
En una célula humana cualquiera, alrededor de 500 bases sufren daño oxidativo cada día.De estas lesiones oxidativas, las más peligrosas son las roturas de doble hebra, ya que son difíciles de reparar y pueden producir mutaciones puntuales, inserciones y deleciones de la secuencia de ADN, así como translocaciones cromosómicas.
Muchos mutágenos se posicionan entre dos pares de bases adyacentes, por lo que se denominan agentes intercalantes. La mayoría de los agentes intercalantes son moléculas aromáticas y planas, como el Bromuro de etidio, la daunomicina, la Doxorubicina y la Talidomida.
Para que un agente intercalante pueda integrarse entre dos pares de bases, éstas deben separarse, distorsionando las hebras de ADN y abriendo la doble hélice. Esto inhibe la Transcripción y la Replicación del ADN, causando toxicidad y mutaciones. Por ello, los agentes intercalantes del ADN son a menudo Carcinógenos: el Benzopireno, las Acridinas, la Aflatoxina y el Bromuro de etidio son ejemplos bien conocidos.Sin embargo, debido a su capacidad para inhibir la replicación y la transcripción del ADN, estas toxinas también se utilizan en Quimioterapia para inhibir el rápido crecimiento de las células cancerosas.
El daño en el ADN inicia una respuesta que activa diferentes mecanismos de reparación que reconocen lesiones específicas en el ADN, que son reparadas en el momento para recuperar la secuencia original del ADN. Asimismo, el daño en el ADN provoca una parada en el Ciclo celular, que conlleva la alteración de numerosos procesos fisiológicos, que a su vez implica síntesis, transporte y degradación de proteínas (véase también Checkpoint de daños en el ADN). Alternativamente, si el daño genómico es demasiado grande para que pueda ser reparado, los mecanismos de control inducirán la activación de una serie de rutas celulares que culminarán en la muerte celular.
Bibliografía
- Clayton, Julie. (Ed.). 50 Years of DNA, Palgrave MacMillan Press, 2003. ISBN 978-1-4039-1479-8.
- Judson, Horace Freeland. The Eighth Day of Creation: Makers of the Revolution in Biology, Cold Spring Harbor Laboratory Press, 1996. ISBN 978-0-87969-478-4.
- Olby, Robert. The Path to The Double Helix: Discovery of DNA, first published in October 1974 by MacMillan, with foreword by Francis Crick; ISBN 978-0-486-68117-7; the definitive DNA textbook, revised in 1994, with a 9 page postscript.
- Ridley, Matt. Francis Crick: Discoverer of the Genetic Code (Eminent Lives) HarperCollins Publishers; 192 pp, ISBN 978-0-06-082333-7 2006.
- Rose, Steven. The Chemistry of Life, Penguin, ISBN 978-0-14-027273-4.
- Watson, James D. and Francis H.C. Crick. A structure for Deoxyribose Nucleic Acid (PDF). Nature 171, 737–738 p. 25 de abril de 1953.
- Watson, James D. DNA: The Secret of Life ISBN 978-0-375-41546-3.
- Watson, James D. The Double Helix: A Personal Account of the Discovery of the Structure of DNA (Norton Critical Editions). ISBN 978-0-393-95075-5.
- Watson, James D. "Avoid boring people and other lessons from a life in science" (2007) New York: Random House. ISBN 978-0-375-41284-4.
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F. and Travers, Andrew A. Understanding DNA, Elsevier Academic Press, 2003. ISBN 978-0-12-155089-9.
Fuentes
- Modelo en 3 dimensiones de la estructura del ADN. Interactivo. Requiere Java.
- Experimento para extraer ADN
- «Descifrar la vida»: Gráfico interactivo
- La Replicación de ADN: características, proteínas que participan y proceso
- Animación en 3D de la Replicación de ADN
- Proceso de extracción de ADN
- Uso del ADN en medicina forense
- Creación del primer genoma artificial
- Construye una molécula de ADN
- El método de secuenciación de Sanger
- El misterioso elfo de Leewenhoek: el recorrido desde el microscopio hasta el ADN en el Museu Virtual Interactivo de la Genética y el ADN