Mononegavirales
| ||||||
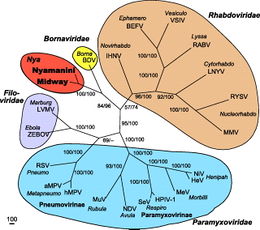
El orden Mononegavirales es el hogar taxonómico de numerosos virus relacionados. Los miembros de la orden que se conoce comúnmente son, por ejemplo, el virus del Ébola, virus Sincitial respiratorio, virus del Sarampión, virus de las Paperas, virus de Nipah, y el virus de la Rabia. Todos estos virus causa una enfermedad significativa en seres humanos. Muchos patógenos muy importantes de animales y plantas no humanos también son miembros de este orden.
Sumario
Estructura
Su genoma es lineal, por lo general no segmentado, de cadena simple, ARN infeccioso de polaridad negativa, no está unido covalentemente a una proteína, la proteína tiene una envueltura de polimerasa dependiente de ARN de genes 5 UTR. Los viriones poseen una envoltura y un nucleocápsido helicoidal con una polimerasa RNA-dependiente (RDRP) y un ARN genómico. El genoma contiene 6-10 genes, aunque un proceso conocido como edición de ARN permite un gran número de productos génicos.
Ciclo de vida
Entrada
Las partículas virales entran a la célula para enlazarse a un receptor de célula superficial ácido siálico e induce la fusión entre la envoltura viral y la membrana celular. En el citoplasma, la partícula se descubre, lanzando el genoma.
Síntesis del mARN
La secuencia genómica es de sentido negativo, esto es que no codifica para proteínas. Las secuencias complementarias deben primero transcribirse geneticamente con una polimerasa ARN dependIente. Esto da inhabilidad al genoma para producir proteínas que requiere el virión en transportar el RDRP con él dentro de la célula. Esto es en contraste con las razas de virus positivas que pueden sintetizar RDRP una vez dentro de la célula. El RDRP lanzado de la partícula viral se une al único promotor de secuencias al tercio final del genoma y comienza la transcripción. El RDRP pausa en los huecos entre cada gene, lanzando el mRNA completado. La traducción puede tanto terminar o continuar transcribiendo el siguiente gene. Esto crea una polaridad en la transcripción, donde los genes cierran el tercio final del genoma, transcribiendo en la máxima abundancia, mientras los del 5º final están al menos listos para ser transcriptos, desde el RDRP hay más oportunidades de terminar. Colocando los genes en orden de abundancia de mayor a menor, el virus rs capaz de usar esta polaridad como una forma de regulación transcripcional. En consecuencia, muchos genomas comienzan con el gene para la proteína del nucleocápsido y finalizan con el gene para RDRP.
Síntesis de proteína y de genoma
Una vez que la producción de ARNm ha comenzado, el sistema de traducción de la proteína celular es cooptado para producir proteínas virales que se acumulan en el citoplasma. En algún momento, posiblemente determinada por la concentración de la proteína de la nucleocápside, las moléculas RDRP transcribir el genoma viral comienzan ignorando las secuencias de la brecha entre los genes y producen de larga duración, antigenomas de cadena positiva. Estos a su vez se transcriben en las copias del genoma viral de cadena negativa.
Ensamblaje y salida
Las proteínas y genomas virales recién sintetizadas se auto-ensamblan y se acumulan cerca del interior de la membrana celular. Los viriones brote fuera de la célula, la obtención de un sobre de la membrana celular cuando salen. La nueva partícula viral infecta a otra célula para repetir el ciclo.
Evolución
Al igual que con otros virus de ARN que no tienen un intermedio de ADN, los miembros de los Mononegavirales son capaces de evolucionar a un ritmo rápido debido a la ausencia de la capacidad de corrección de lectura en la enzima RDRP. Una alta tasa de mutación se produce en la producción de nuevos genomas (hasta 1 por 1000 bases).