Degradación de Edman
| ||||||
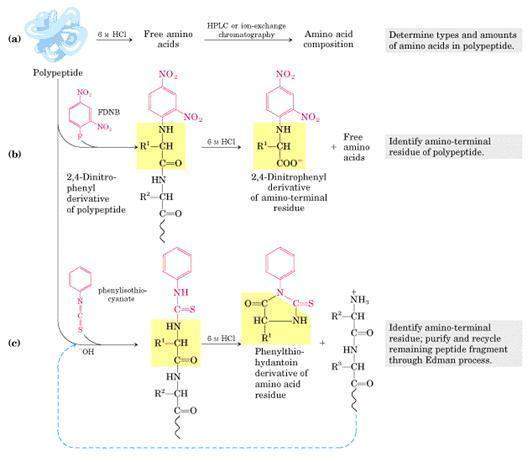
La degradación de Edman. Es un método de secuenciación de aminoácidos en un péptido, donde el residuo amino terminal se etiqueta y se separa del péptido sin afectar a los enlaces peptídicos entre los otros residuos.
Historia
Pehr Victor Edman a traves de sus ensayos logró reconocer que las proteínas son entidades con una estructura, una masa y una carga eléctrica definidas. Su investigación básica en Estocolmo, en el Instituto Karolinska, como parte de su trabajo para su tesis doctoral, el cual se vio interrumpido por la guerra, y tras servir en el ejército sueco durante la Segunda Guerra Mundial continuó sus trabajos.
En 1947, le concedieron un premio para ir al Rockefeller Institute of Medical Research en la prestigiosa Universidad de Princeton donde realizó sus trabajos de purificación y caracterización de la Angiotensina de sangre bovina.
Edman determinó detalladamente la estructura de las proteínas y con su método consiguió dar lugar a una explicación estructural de sus diversas actividades biológicas. Aisló de la sangre bovina una proteína pequeña que regula la presión arterial (Angiotensina), lo que le permitió conocer que las proteínas están constituidas por pequeños “bloques o ladrillos”: los aminoácidos.
Para dar una explicación estructural a las diversas actividades biológicas de las proteínas encontró un método que le permitió la determinación de la estructura de las mismas y desempeñó una serie de reacciones basadas en el acoplamiento del fenilisotiocianato (PITC) con una proteína purificada y el uso de hidrólisis ácida. Esta serie de reacciones le permitieron analizar las proteínas de forma secuencial, aminoácido por aminoácido, y así preservar la información lineal estructural necesaria para interpretar su actividad biológica.
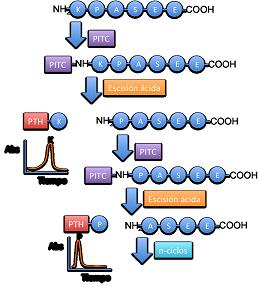
El procedimiento de la degradación de Edman o secuenciación de Edman marca y elimina sólo el residuo N-terminal de un polipéptido, dejando el resto de enlaces peptídicos intactos. Para ello, se hace reaccionar el péptido con PITC eliminándose el residuo N-terminal en forma de derivado de fenilhidantoína (PTH). Después de la eliminación e identificación del residuo N-terminal mediante HPLC, el nuevo residuo N-terminal puede ser marcado, eliminado e identificado por repetición de la misma serie de reacciones. Este procedimiento se repite hasta que se ha determinado toda la secuencia.
Esquema de la Secuenciación de Edman
Edman ideó en la Universidad de Lund las condiciones de reacción adecuadas para todos los aminoácidos y la gran mayoría de las clases de proteínas además de minimizar el número de reacciones secundarias no deseadas.
Debido a que se tienen que realizar muchos pasos de reacción y muchas determinaciones, normalmente hoy día éste método se lleva a cabo mediante analizadores automáticos o secuenciadores que mezclan los reactivos en las proporciones adecuadas, separa los productos, los identifica y registra los resultados.
Diseñó en 1961 un instrumento automático llamado “secuenciador de proteína para determinar la estructura de las proteínas mediante el análisis de la secuencia de aminoácidos”, el primer secuenciador automatizado de proteínas.
Edman continuó hasta su muerte trabajando para mejorar este método y así poder determinar cadenas más largas pero con menor material de partida. La secuenciación de proteínas constituyó uno los hitos históricos a tener en cuenta para las personas que trabajan con las mismas ya que sin ella probablemente no se tendría la Proteómica.
En medio básico, se provoca la reacción del extremo amino con fenilisotiocianato para formar un péptido feniltiocarbamilado. A continuación, se aplica un medio ácido para que el tiocarbamoílo forme un ciclo de 5 miembros (feniltiohidantoína) con el carbonilo del enlace peptídico adyacente. De este modo, se obtiene un péptido más corto, con un nuevo extremo amino y la feniltiohidantoína del primer aminoácido.
Limitaciones
En este mecanismo no es posible secuenciar péptidos cuyo extremo N-terminal se encuentre bloqueado (grupo amino acetilado, formilado, etc). Alrededor del 50% de las proteínas tienen el extremo amino bloqueado, bien por naturaleza o por su preparación, en tampones con sustancias susceptibles de reaccionar con este: (cianato, ácido acrílico, aldehído).
Otra limitación de la degradación de Edman es que el rendimiento no es del 100% y por tanto cuando ya se llevan hechos muchos ciclos (más de 50 aminoácidos analizados) se obtienen errores.Este procedimiento se repite hasta que se ha determinado toda la secuencia.