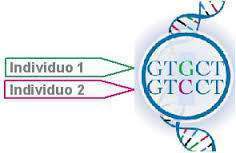
Polimorfismo de nucleótido simple
| ||||||
Polimorfismos de nucleótido único o simple (del inglés Single Nucleotide Polymorphisms, SNPs), (se pronuncia como snips). Son la forma más sencilla y más común de polimorfismo genético ya que consisten en el cambio de un solo nucleótido en el contexto de una secuencia genética. Se considera que ellos determinan la mayor parte de la variabilidad genética entre los individuos, causando muchas de las diferencias fenotípicas (observables) en ellos.
Sumario
Ubicación en el genoma
Un SNP es una variación en la secuencia de ADN que afecta a una sola base nitrogenada de una secuencia del genoma: adenina (A), timina (T), citosina (C) o guanina (G). Algunos autores consideran que cambios de unos pocos nucleótidos, como también pequeñas inserciones y deleciones (indels) pueden ser consideradas como SNP, si una de estas variaciones se da al menos en un 1% de la población; si no se llega al 1% no se considera SNP y sí una mutación puntual.
Los SNPs se distribuyen de manera heterogénea por todo el genoma y se encuentran tanto en las regiones codificantes (exones) como no codificantes (intrones y región promotora) de los genes así como en las zonas del genoma en donde no asientan genes conocidos (a veces llamado “ADN basura”).
Se han descrito varios millones de SNP distribuidos por todos los cromosomas humanos, animales y de plantas. Se estima que su frecuencia media, aunque con grandes variaciones según la especie, cromosomas y regiones dentro de ellos, es de 1 SNP cada 500-1000 nucleóticos, o quizás menos, de solo uno cada 100–300 pb (pares de bases).
Los SNP que se localicen dentro de una secuencia codificante pueden modificar o no la cadena de aminoácidos que producen, se llama SNP no-sinónimos a los primeros y SNP sinónimo (o mutación silenciosa) a los segundos. Los SNP que se encuentren en regiones no codificantes pueden tener consecuencias en el proceso de traducción, sobre todo en procesos como el splicing, la unión de factores de transcripción o modificando la secuencia de ARN no codificante. Aunque pueden estar tanto en regiones codificantes como en regiones intrónicas o intergénicas, las codificantes son las que tienen más impacto sobre la función de una proteína, pero todas pueden estar relacionadas con una enfermedad.
Importancia
Los SNPs se consideran una forma de mutación puntual que ha sido lo suficientemente exitosa evolutivamente para fijarse en una parte significativa de la población de una especie. Este tipo de polimorfismo tiene una gran importancia biológica, ya que determinan la mayor parte de la variabilidad genética de los individuos, causando muchas de las diferencias fenotípicas (observables) de los mismos.
Se cree que se trata de mutaciones puntuales acaecidas en diferentes momentos de la historia evolutiva de la especie y que en su momento fueron estabilizadas en el genoma humano por conferir algún tipo de ventaja adaptativa al medio en ese momento (por ejemplo, mayor capacidad metabólica o de detoxificación al ingerir un nuevo tipo de alimento).
Desde el punto de vista médico también tienen una gran relevancia pues las diferentes alternativas de un determinado SNP pueden determinar, por ejemplo, diferente propensión a desarrollar una enfermedad, una mayor agresividad clínica de la misma o diferencias en la forma de responder al tratamiento.
Los SNPs constituyen hasta el 90% de todas las variaciones genómicas humanas, y aparecen cada 1,300 bases en promedio, a lo largo del genoma humano. Dos tercios de los SNP corresponden a la sustitución de una citosina (C) por una timina (T). Estas variaciones en la secuencia del ADN pueden afectar a la respuesta de los individuos a enfermedades, bacterias, virus, productos químicos, fármacos, etc.
Detección y utilidad en estudios genéticos en plantas
El tipo de polimorfismo de los SNPs puede detectarse por varios métodos y dada su gran importancia biológica, se han utilizado con éxito en varios cultivos. Los SNPs pueden aislarse a partir de ADNc, ARN mensajero y ESTs o secuencias expresadas y marcadas (Expressed Sequence Tags, ESTs).
Los métodos de detección basados en la PCR son los preferidos por su eficiencia y rapidez. La técnica de SSCP conocida como polimorfismo conformacional de cadena simple (Single Strand Conformational Polymorphism, SSCP), es la recomendada para detectar los SNPs por su capacidad para identificar mutaciones puntuales. Esta técnica se basa en la migración diferencial del ADN y es capaz de distinguir cambios de pocos nucleótidos en una secuencia de más de 1 Kb.
Además de su uso en humanos, los SNPs tienen una aplicación importante en el mejoramiento vegetal, las prácticas agronómicas y otras investigaciones, sobre todo en los países en vías de desarrollo; encaminadas a definir la estructura génica y relaciones funcionales y evolutivas. Esto incluye análisis fitogenéticos, el empleo de la selección asistida por marcadores (SAM), el mapeo de QTLs o loci para caracteres cuantitativos (del inglés, Quantitative Trait Loci), la selección de genoma y los estudios de asociación. A partir de las secuencias públicas de las ESTs y la existencia de los SNPs se puede buscar asociación de marcadores moleculares con características de interés para los mejoradores.
Entre los cultivos con mayor aplicación de los SNPs están el maíz (Zea mays L.), arroz (Oriza sativa L.) y la caña de azúcar (Saccharum officinarum L.). Dadas las ventajas para la utilización de los SNPs, basados en técnicas de PCR (reacción en cadena de la polimerasa), cabe esperar un incremento sustancial en sus aplicaciones en los análisis de asociación de genoma completo (genome-wide association studies, GWAS).
Recientemente, se han informado sobre avances en el uso de SNPs en estudios relacionados con la cianogénesis en yuca (Manihot esculenta Crantz), el metabolismo del almidón y las vías relacionadas con el estrés, o la resistencia a la bacteriosis vascular.
En Cuba el primer informe sobre aplicación de los SNPs en la mejora asistida de un cultivo de importancia económica, ha sido en yuca, para la búsqueda de marcadores moleculares asociados al porcentaje de materia seca, con una contribución importante al esquema de mejora genética del cultivo y el primer informe a nivel internacional en ese tema.
Enlaces externos
- dbSNP, la mayor base de datos que existe sobre los diferentes tipos de SNP. Auspiciada por el Centro Nacional de Información Biotecnológica, (en inglés, NCBI)
- International HapMap Project. Un recurso público que ayuda a los investigadores a encontrar genes asociados con enfermedades humanas y respuesta a fármacos.
- Restriction HomePage. Conjunto de herramientas para tratamiento del DNA con enzimas de restricción y detección de SNP, incluyendo diseño de cebadores mutagénicos.
- Seattle SNP site. Base de datos curada de SNP.
- SNPStats. Herramienta web para el análisis de estudios de asociación genética.
- The SNP Consortium LTD. Búsqueda de SNP.
Referencias
- Beovides, Y. (2014). Caracterización de la variabilidad genética e identificación de marcadores moleculares asociados al porcentaje de materia seca en accesiones cubanas de yuca (Manihot esculenta Crantz). Tesis de Doctorado, Facultad de Biología, Universidad de la Habana Cuba, 156 p.
- Contreras E y C López (2011) Identificación de polimorfismos en RXam2, un gen candidato de resistencia a la bacteriosis vascular de yuca. Rev Colomb Biotecnol XIII(2): 63-69.
- Deschamps S y MA Campbell (2010) Utilization of next-generation sequencing platforms in plant genomics and genetic variant discovery. Mol Breed 25: 553–570.
- Edwards D; JW Forster; NOI Cogan; J Batley; D Chagne’ (2010) Single nucleotide polymorphism discovery. In: Oraguzie NC; Rikkerink EHA; Gardiner SE; De Silva HN (eds) Association mapping in plants. Springer, New York, p. 53–76.
- Huang X; T Sang; Q Zhao; X Wei; Q Feng; Y Zhao; C Li; C Zhu; T Lu; Z Zhang; M Li; D Fan; Y Guo; A Wang; L Wang; L Deng; W Li; Y Lu; Q Weng; K Liu; T Huang; T Zhou; Y Jing; W Li; L Zhang; ES Buckler; Q Qian; Q Zhang; J Li y B Han (2010) Genome-wide association studies of 14 agronomic traits in rice landraces. Nature Genetics 42(11): 961-967.
- López C; B Piegu; R Cooke; M Delseny; J Thome y V Verdier (2005) Using cDNA and genomic sequences as tools to develop SNP strategies in cassava (Manihot esculenta Crantz). Theor Appl Genet 110: 425-431.
- Owusu K (2008) Expressed sequence tags (ESTs) and single nucleotide polymorphisms (SNPs): Emerging molecular marker tools for improving agronomic traits in plant biotechnology. Afr J Biotech 7(4): 331-341.