ATP Sintetasa
| ||||||
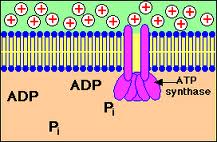
ATP Sintetasa. Complejo enzimático de la membrana interna de la mitocondria y de la membrana tilacoide del cloroplasto, a través del cual fluyen los protones a favor del gradiente establecido en la primera etapa del acoplamiento quimiosmótico; el sitio de formación de ATP a partir de ADP y fosfato inorgánico durante la fosforilación oxidativa y la fotofosforilación.
Función
El complejo ATP sintetasa es una enzima encargada de sintetizar Adenosín Trifosfato (ATP) a partir de ADP y un grupo fosfato, merced a la energía suministrada por un flujo de protones, de acuerdo con la hipótesis quimiosmótica de Mitchell. La ATP sintetasa se puede imaginar como un motor molecular que produce una gran cantidad de ATP cuando los protones fluyen a través de ella. La tasa de síntesis es grande, el organismo humano en fase de reposo puede formar unas 1021 moléculas de ATP por segundo.
La ATP es un compuesto de alto contenido energético utilizado en casi todos los procesos bioquímicos del cuerpo humano. La ATP sintetasa se nos muestra como un fascinante y minúsculo motor rotatorio que trabaja con una relación de eficiencia cercana al 100% y cuya función resulta esencial para cualquier proceso biológico, por lo que su presencia imprescindible y su indudable complejidad irreducible lo convierten en un elemento que ha despertado desde su conocimiento detallado, profundas sospechas de haber sido diseñado.
Estructura
La ATP sintasa tiene un diámetro de 10 nm, y es el complejo más pequeño identificado hasta ahora. Trabaja con un grado de efectividad cerca al 100 por ciento.
Esta enzima está formada por dos principales complejos. Una anclada a la membrana mitocondrial interna o al tilacoide llamada F0 (CF0 en caso de los tilacoides) y otra que sobresale por la cara interna de la estructura llamada F1 (CF1 en caso de los tilacoides).
El componente F0 es el motor impulsado por protones. Es conocida como la fracción sensible a la oligomicina está formada por las subunidades a, b2 y c10-14. Las subunidades c forman el “anillo c”, que rota en sentido horario en respuesta al flujo de protones por el complejo. Las dos proteínas b inmovilizan el segundo complejo F1, que está orientada hacia la matriz mitocondrial. Por interacciones electrostáticas, se asocia a F1 a Fo.
F1 está formada por las subunidades α3, β3, γ, δ y ε. La parte principal del complejo F1 está formado por tres dímeros αβ, esta unidad tiene forma de hexámero. La actividad catalitítica de este hexámero está localizada en las subunidades β. Las subunidades γ y ε están unidas al anillo c, y giran con él. Cada rotación de 120º de la subunidad γ induce la aparición de cambios de conformación en los centros catalíticos de las unidades β del los dímeros αβ, provocando la alteración de los centros de fijación de los nuceótidos situado en β. El hexámero α3 y β3 finalmente libera el ATP.
La subunidad Fo se conforma de once subunidades diferentes a1 y c10, estas subunidades forman un canal de protones central y finalmente la subunidad b2OSCP1 (Oligomycin Sensitiviy Conferring Protein) enlazan las unidades F1 y Fo.
Inhibidores
La oligomicina se fija en el tallo de la ATP sintasa, inhibiendo el canal de protones, por tanto previniendo el reingreso de protones en la matriz mitocondrial. Como los gradientes de pH y eléctricos no se pueden disipar en presencia de la oligomicina, el transportador de protones se detiene por la dificultad para el bombeo de más protones contra los gradiententes muy inclinados. Causa una acumulación de protones en el espacio intermembrana.