Intrón
| ||||||
Intrón. Fue descubiertos por Phillip Allen Sharp y Richard J. Roberts, ganando con ellos el Premio Nobel de Fisiología o Medicina en 1993.
Sumario
Origen
El término intrón fue introducido por el bioquímico estadounidense Walter Gilbert en 1978. Los intrones pueden representar un sitio alternativo de splicing, pudiendo dar diferentes tipos de proteínas. El control del splicing está regulado por una amplia variedad de señales moleculares. Los intrones también pueden contener “información antigua”, es decir, fragmentos de genes que probablemente se expresaban pero que actualmente no se expresan. Se ha especulado con el origen de los intrones y que tal vez venían desde transposones, por su habilidad para insertarse dentro de secuencias codificantes. Esta relación está basada en que el sistema para sacar los intrones de la secuencia del ARN y algunos transposones es similar, los intrones tienen unas secuencias repetidas a los lados que les permiten la circularización y la eliminación por unas endonucleasas relacionas con las de transposones.
Fisiología
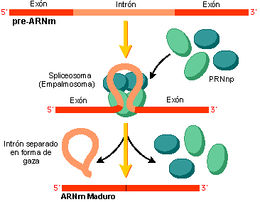
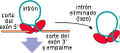
Los intrones son las secuencias de ADN o ARN secuencias que separan los exones. Ambos se transcriben de ADN a ARN, y de ese transcrito primario (o pre-ARNm) serán eliminados los intrones durante la maduración del ARN. Estas secuencias no contienen información propiamente dicha sobre los aminoácidos que darán lugar a la proteína, sin embargo son importantes funcionalmente, puesto que en parte son responsables por ejemplo, del barajado de exones. Los intrones son característicos de eucariotas, si bien se han encontrado algunos en procariotas, no representan una parte tan importante de su genoma. La frecuencia de los intrones en el genoma es muy variable dependiendo del organismo que estudiemos. Por ejemplo, los vertebrados superiores como el ser humano o las vacas tienen un porcentaje muy elevado de intrones y secuencias que no codifican proteínas. En bacterias y archeas son más raros pero también hay. Parece haber algún tipo de relación entre el número de intrones y en general de las secuencias no codificantes y la complejidad evolutiva de una especie. En contraste los genomas de mitocondriasno tienen ningún intrón. De hecho, las mitocondrias tienen algunos genes con la secuencia de bases solapante, algo insólito en eucariotas. Los intrones son eliminados de la secuencia de ARN mensajero por el proceso denominado splicing alternativo, que engloba tanto la eliminación del intrón mediante proteínas especializadas como la unión posterior de los exones que quedan sueltos.
Clasificación
Actualmente se reconocen cuatro clases de intrones:
Intrones del grupo I
- Se auto-procesan in vitro aunque algunos necesitan la intervención de la proteína que está codificada por el ORF del intrón: las madurasas. Algunas de ellas también tienen actividad retrotranscriptasa o endonucleasa que sirven para la transposición y el homing del intrón.
- Requieren un cofactor externo que es un nucleósido o nucleótido de guanina (guanosina, GMP, GDP o GTP) en el que el 3’OH actúa como nucleófilo.
- No se necesita fuente de energía porque se utiliza la energía de un tipo particular de transesterificación: la transfosforilación.
- La unión del cofactor externo desencadena el proceso, sin formación de lazos (lariat).
- No existen secuencias de consenso claras para producir el ayuste o reconocer los bordes del intrón.
- Se han identificado unas pequeñas secuencias que suelen ser importantes, directa o indirectamente, para el procesamiento. Su conservación sugiere que un ancestro común migró a la mitocondria desde el núcleo, o viceversa. Estas secuencias forman dos estructuras típicas:
- una de ellas está formada por el apareamiento de 6-7 nt entre las secuencias P y Q de 10 nt de longitud (P4 en la figura). En el caso de intrones mitocondriales se denominan secuencias «9L»
- La otra estructura la forma el apareamiento de 5 nt entre los elementos R y S de 12 nt (P7 en la figura). En el caso de intrones mitocondriales, reciben el nombre de «2».
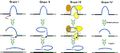
Intrones del grupo II y III
- Se autoprocesan in vivo aunque algunos también necesitan madurasas, y utilizando la energía de la transfosforilación
- No necesitan un cofactor externo ya que utilizan el 2’OH de un residuo de A del propio intrón
- Por utilizar como cofactor un nucleótido del intrón, dan lugar a un intermedio en forma de lazo (lariat).
- Aunque la secuencia de nucleótidos no está conservada, presentan una estructura secundaria formada por 6 horquillas denominadas d1 a d6. En d6 se encuentra la A que comenzará el ataque nucleófilo. El centro catalítico del intrón viene definido por d1 y d5.
- Con frecuencia se comportan como elementos móviles, por lo que se les considera un tipo particular de retrotransposón. Para saltar a un nuevo lugar (homing) emplean tanto endonucleasas como retrotranscriptasas codificadas por el propio intrón u otros intrones del mismo grupo.
Su estructura y su mecanismo de ayuste se parecen mucho a los intrones nucleares, por lo que se les cree relacionados evolutivamente. Existe un subgrupo de estos intrones que carecen de los dominios d2 a d4 y se denominan frecuentemente intrones del grupo III, que no hay que confundir con los intrones nucleares (véase a continuación).
Intrones nucleares, spliceosomales o intrones del grupo IV
Es el más numeroso, con intrones presentes en la mayoría de los mRNA nucleares eucarióticos. El ayuste es similar al del grupo II, pero necesita la intervención un conjunto muy numeroso de proteínas y RNA para formar el ayustosoma —con gasto de ATP—. Todavía no se ha encontrado ninguno de este tipo en procariotas. Agrupa los intrones presentes en los tRNA. A diferencia de los tres anteriores, se necesitan endonucleasas para eliminar el intrón y ligasas para unir los exones. Se han encontrado en los eucariotas y en las arqueas.
¿Dónde se encuentran?
Se encuentran común en eucariotas multicelulares, tales como seres humanos. Son menos comunes en eucariotas unicelulares, tales como levadura, e incluso más raro en bacterias. Se ha sugerido que el número de intrones que los genes de un organismo contienen está relacionado positivo con su complejidad.
Galería
Referencias
- Saladrigas V, Claros G (2002): «Vocabulario inglés-español de bioquímica y biología molecular »(1.ª entrega) Panace@ III (9-10): 13-28. Vocabulario completo en BioROM.
- Gilbert, Walter (1978): «Why genes in pieces.» Nature 271(5645): 501. Plantilla:DOI
- Roy, Scott William & Gilbert, Walter (2006): «The evolution of spliceosomal introns: patterns, puzzles and progress.» Nature Reviews Genetics 7: 211-221. Plantilla:DOI PDF fulltext
Fuente
- Artículo sobre Intrón. Disponible en el sitio web:Enciclopedia. Consultado el 19 de abril de 2017.
- Artículo sobre Intrón. Disponible en el sitio web:Biología. Consultado el 19 de abril de 2017.
- Artículo sobre Intrón. Disponible en el sitio web:Unav. Consultado el 19 de abril de 2017.
- Artículo sobre Intrón. Disponible en el sitio web:Biorom. Consultado el 19 de abril de 2017.