Mapeo de asociación
| ||||
Mapeo de asociación. Método de mapeo de loci para caracteres cuantitativos o QTLs (del inglés, Quantitative Trait LociTexto en cursiva) que aprovecha el vínculo histórico del DL en la asociación entre los fenotipos (características visibles) y los genotipos (constitución genética de los organismos). A menudo referido como desequilibrio de ligamiento (DL), se basa en la asociación no aleatoria entre los marcadores y las características de interés.
Sumario
Generalidades
Tras los estudios de genética clásica sobre la transmisión de caracteres independientes por parte de Mendel que condujeron en el siglo XIX a la formulación de sus leyes, otros investigadores, William Bateson y R. C. Punnet, encontraron en 1911 una desviación a este tipo de herencia al estudiar la segregación de cruzamientos entre dos líneas de flores que diferían en dos caracteres dados. Aunque la base física de esta peculiaridad se desconocía en la época, dicha anomalía se debe a que aquellos caracteres se encontraban acoplados físicamente en el cromosoma: esto es, se encontraban ligados, a diferencia de los estudiados por Mendel, que se encontraban en cromosomas distintos.De esta manera, la tasa de recombinantes producidos tras el cruce de los parentales era menor a la esperada, debido a que, por cercanía física en la hebra de ADN, la probabilidad de que se produjese un evento de recombinación (un quiasma) era pequeña, e inversamente proporcional a la distancia entre los dos loci codificantes de esos caracteres. En cambio, en el modelo inicial de Mendel, la probabilidad de recombinación, de 0,5, expresaba la transferencia aleatoria de uno u otro alelo de cada carácter debido a la transferencia al azar hacia el gameto de uno u otro cromosoma durante la meiosis.El mapeo de asociación (MA) busca identificar marcadores ligados a variaciones fenotípicas en un carácter de interés a partir de una muestra de individuos, sobre la base del desequilibrio de ligamiento observado en poblaciones no estructuradas como pueden ser poblaciones naturales (humanas, animales o de plantas), colecciones de germoplasma, líneas o variedades elite de un programa de mejoramiento, etc.
Importancia teórico-práctica
El desarrollo de estrategias para el mapeo de asociación en plantas y animales, está entre los aportes de mayor impacto en el mejoramiento genético, acortando significativamente el tiempo necesario para obtener nuevas variedades y razas. Particularmente útil son las aplicaciones del mapeo de asociación en humanos Otra alternativa eficiente para el mapeo de asociación que ha alcanzado gran auge en los inicios del siglo XXI es los estudios de asociación de genoma completo (genome-wide association studies, GWAS), el cual no requiere conocer información previa del genoma de los organismos en estudio. La ventaja del mapeo de asociación es que puede mapear los rasgos cuantitativos con una alta resolución ya que esa vía es estadísticamente muy poderosa. Sin embargo, el mapeo de asociación también requiere un conocimiento amplio del genoma del organismo de interés, y es por consiguiente difícil de realizar en especies que no se han estudiado bien o no se conocen bien aún sus genomas. No obstante, algunos autores consideran que la búsqueda de marcadores genéticos puede acelerarse por sendas que no requieren desarrollar el mapeo de las poblaciones, o sin conocer completamente el genoma de una especie, con algunas variantes del mapeo de asociación y principalmente en plantas.
Mapeo de asociación en plantas
En plantas, el mapeo por asociación es una alternativa al mapeo genético y consiste en que, a partir del polimorfismo en genes candidatos dentro de un grupo de individuos particulares, se puede establecer una asociación de ellos con un fenotipo de interés. Estos estudios tienen un amplio uso en las estrategias de mejora genética de los cultivos.
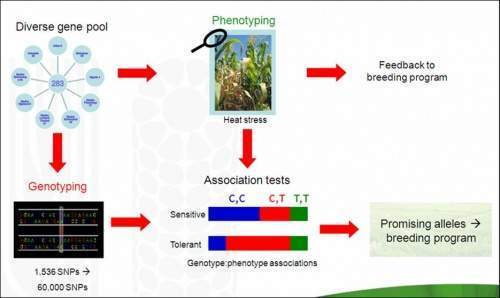
En los últimos años se han desarrollado estrategias basadas en el mapeo por asociación como una alternativa al mapeo genético, y a partir del polimorfismo en genes candidatos dentro de un grupo de individuos particulares, no necesariamente poblaciones o colecciones producto de cruzamientos dirigidos, se puede establecer una asociación con el fenotipo.Cuando existe desequilibrio de ligamiento entre un marcador molecular y un locus desconocido que controla un fenotipo de interés, haplotipos específicos como los SNPs o marcadores de alelos pueden ser asociados con los valores fenotípicos medidos en la colección de individuos en estudio. No obstante, para detectar la asociación entre el marcador de ADN y el factor desconocido que controla el carácter cuantitativo como el PMS en yuca, el marcador tendrá que estar física y fuertemente ligado, tanto que el ligamiento sea posible más allá de distancias grandes entre pares de loci. Adelantos tecnológicos en la secuenciación del ADN, así como, el desarrollo de métodos paralelos para su genotipado y el desequilibrio de ligamiento de alta resolución, basado en el mapeo de asociación de polimorfismos del nucleótido, facilitan los análisis directos de diversidad del nucleótido y el descubrimiento de SNP en varios eucariontes.
Importancia en el mejoramiento genético
La búsqueda de marcadores estrechamente ligados a genes de interés garantiza que la presencia del alelo deseado del gen pueda inferirse por la presencia de un alelo determinado del marcador. Este enfoque es particularmente útil en caracteres de baja heredabilidad, aquellos que su evaluación sea destructiva, difícil de llevar a cabo en condiciones de campo o donde se requiere de mucho tiempo o recursos.Por tales razones, este tipo de alternativas son valiosas en plantas que presentan un ciclo de vida largo, baja producción de semillas y por tanto la realización de cruces exige largos períodos. Para identificar asociación entre el polimorfismo molecular y el fenotipo de caracteres cuantitativos a través del mapeo de asociación del genoma completo, se necesitan densidades muy altas de marcadores de ADN, lo que se puede lograr con el empleo de los SNPs.
También son de ayuda en la selección de padres donantes y en la introgresión de caracteres de interés desde parientes silvestres o cultivados.
Aplicación en la mejora asistida
Son interesantes los avances que se logran con el uso de esta tecnología en la búsqueda de resistencia al estriado marrón de la yuca (cassava brown streak disease, CBSD) a partir de cultivares africanos y mediante la aplicación de la técnica de polimorfismo de nucleótido simple (SNP). Esa técnica también fue útil en Cuba para la identificación de marcadores moleculares asociados a los más bajos porcentajes de materia seca en yuca. Al mismo tiempo, estudios relacionados con la cianogénesis en yuca, el metabolismo del almidón y las vías relacionadas con el estrés o la resistencia a la bacteriosis vascular, han sido posibles gracias al mapeo de asociación.Aunque hay características que están gobernadas por varios genes, es posible encontrar marcadores de tipo SNP relacionados con un fenotipo de interés. Así sucede con la resistencia a patógenos en varias especies de plantas, donde cambios puntuales en un gen explican las diferencias entre cultivares resistentes y susceptibles. Lo mismo ocurre con la detección de varios genes en arroz y otros responsables de la acumulación de carbohidratos y ácido abscísico durante la floración del maíz bajo estrés por sequía.En arroz, algunos investigadores analizaron la asociación de 17 genes de síntesis de almidón con 43 marcadores específicos utilizando el mapeo de asociación. Igualmente un estudio de secuenciación de 18 genes responsables de la síntesis de almidón en representación de siete enzimas, incluidas SS y SBE, permitió identificar otros 31 SNPs asociados a la textura y la calidad de cocción del arroz.
Tendencias actuales
Con el desarrollo de la biología molecular y la genómica, se abren nuevos horizontes y mayor velocidad en los descubrimientos relacionados con el mapeo de asociación. La implementación de nuevas herramientas basadas en el uso de marcadores moleculares ha revolucionado las investigaciones en plantas, animales y en humanos.Los marcadores moleculares asociados genéticamente a una característica específica, son de especial interés en programas de mejoramiento vegetal debido a que permiten un rápido monitoreo de grandes números de plantas en una temprana edad del desarrollo, pues ello facilita identificar individuos que contengan el carácter.
El mapeo por asociación para identificar marcadores relacionados con caracteres de interés a partir de genes candidatos de diversas especies, es un proceder que ha ganado importancia en las tendencias del mejoramiento genético actual y los programas dirigidos a estudiar las enfermedades y mejorar la salud humana.
Bibliografía
- Beovides, Y (2014) Caracterización de variabilidad genética e identificación de marcadores moleculares asociados a porcentaje de materia seca en yuca (Manihot esculenta Crantz). Tesis de Doctorado, 146p.
- Berovides V (2002) Medida de la variabilidad genética en poblaciones naturales. En: L. Tagle (ed), Marcadores moleculares: Nuevos horizontes en la genética y la selección de plantas. Cap. 5, Ed. Félix Varela, La Habana, p. 120-146.
- Ceballos H; P Kulakow y C Hershey (2012) Cassava Breeding: Current Status, Bottlenecks and the Potential of Biotechnology Tools. Trop Plant Biol 5: 73-87.
- Coto O y MT Cornide (2002) Principales aplicaciones de los marcadores moleculares. En: L Tagle (ed), Marcadores Moleculares. Nuevos horizontes en la genética y la selección de plantas. Cap. 4, Ed. Félix Varela, La Habana, p. 92-110.
- Gebhardt C; A Ballvora; B Walkemeier; P Oberhagemann y K Schuler (2004) Assessing genetic potential in germplasm collections of crop plants by marker-trait association: a case study for potatoes with quantitative variation of resistance to late blight and maturity type. Mol Breed 13: 93–102.
- Gibson, G. y S.V. Muse (2009) A Primer of Genome Science. MA: Sinauer Associates.
- Griffiths, AJF (2007) Genética. Editorial Médica Panamericana, 849p
- Gupta PK; S Rustgi YP y L Kulwal (2005) Linkage disequilibrium and association studies in higher plants: present status and future prospects. Plant Mol Biol 57(4): 461–485.
- Hall D; C Tegstrom y PK Ingvarsson (2010) Using association mapping to dissect the genetic basis of complex traits in plants. Brief Funct Genomics 9(2): 157-165.
- Hospital F (2009) Challenges for effective marker-assisted selection in plants. Genética 136(2): 303-310.
- Hurtado P; JP Gutiérrez; I Moreno; E Barrera y M Fregene (2006) Population structure, phenotypic information and association studies in long generation crops. In: Project IP3: improving cassava for developing Word. Chapter 12, (a GCP Project) p. 60-96.
- Kharabian-Masouleh A; DLE Waters; RF Reinke; R Ward y RJ Henry (2012) SNP in starch biosynthesis genes associated with nutritional and functional properties of rice. Sci Rep 2: 557.
- Kolkman JM; ST Berry; AJ León; MB Slabaugh; S Tang; W Gao; DK Shintani; JM Burke y ST Knapp (2007) Single Nucleotide Polymorphisms and Linkage Disequilibrium in Sunflower. Genetics 177 (1): 457-468.
- Lander ES y NJ Schork (2006) Genetic dissection of complex traits. Focus 4: 442-458.
- Nussbaum, R.L.; R.R. McInnes y H.F. Willard (2007) Genetics in Medicine. Philadelphia, PA: Saunders Elsevier.
- Ojulong H; MT Labuschangne; L Herselman y M Fregene (2008) Introgression of genes for dry matter content from wild cassava species. Euphytica 164: 163-172.
- Rosyara U.R., J.L. Gonzalez-Hernandez, K.D. Glover, K.R. Gedye y J.M. Stein (2009) Family-based mapping of quantitative trait loci in plant breeding populations with resistance to Fusarium head blight in wheat as an illustration Theor Appl Gen 118:1617-1631.
- Setter TL; J Yan; M Warburton; JM Ribaut; Y Xu; M Sawkins;ES Buckler;Z Zhang y MA Gore (2011) Genetic association mapping identifies single nucleotide polymorphisms in genes that affect abscisic acid levels in maize floral tissues during drought. J Exp Bot 62 (2): 701-716.
- Vásquez A y C López (2012) Identificación de polimorfismos en genes candidatos de resistencia en yuca (Manihot esculenta Crantz). Acta Agronómica 61(2): 133-142.
- Yan CJ; ZX Tian; YW Fang; YC Yang; J Li; SY Zeng; SL Gu; CW Xu; SZ Tang y MH Gu (2010) Genetic analysis of starch paste viscosity parameters in glutinous rice (Oryza sativa L.). Theor Appl Genet 122(1): 63-76.
- Yu, J., J.B.Holland, M.D. McMullen y E.S. Buckler (2008) Genetic design and statistical power of nested association mapping in maize, Genetics 178 (1): 539–551.
