Geminivirus
| ||||||
Los Geminivirus constituyen el grupo de más rápido crecimiento quizás de todos los virus de plantas. Están compuestos por ácido desoxirribonucleico (ADN) y una proteína que conforma la cubierta. El nombre (gemini=gemelo) se debe a la estructura de su partícula, compuesta por dos cubiertas icosaédricas unidas por una de sus caras (Hilje, 1996; ICTV Database, 2002b). Los dos icosaedros son ensamblados juntos, los pentámeros son removidos. Cada partícula geminante contiene 110 subunidades proteicas capsídicas de 29-30 kd y una molécula de ADNss (sentido del virión).
Los geminivirus y Bemisia tabaci previamente coexistieron durante muchas décadas en América Latina sin afectar a especies de plantas cultivables. Hoy América Latina es la región más afectada en cuanto a número total de geminivirus aparecido, número de cultivos afectados, pérdidas en el rendimiento y áreas agrícolas devastadas por este patógeno (Morales y Anderson, 2001).
En la actualidad 5 millones de hectáreas de tierras destinadas a la agricultura primaria en 20 países están bajo el ataque de 30 geminivirus diferentes.
Sumario
- 1 Clasificación de geminivirus
- 2 Sintomatología
- 3 Rango hospedante y transmisión
- 4 Efectos citopáticos y localización en los hospedantes
- 5 Distribución y daños
- 6 Organización genómica
- 7 Proteínas virales
- 8 Replicación viral
- 9 Estudio y diagnóstico
- 10 Importancia económica y control
- 11 Referencias
- 12 Fuentes
Clasificación de geminivirus
Las primeras agrupaciones de la familia Geminiviridae fueron hechas basándose en los síntomas que provocan, posteriormente se clasificaron en tres subgrupos teniendo en cuenta su organización genómica (monopartita o bipartita), la planta hospedante (monocotiledónea o dicotiledónea) y el insecto vector (saltahojas o mosca blanca).Existen otras dos propuestas, en el primer sistema se definen dos subfamilias dentro de la familia Geminiviridae: La Alphageminivirinae (contiene al subgrupo I) y la Betageminivirinae (contiene a los subgrupos II y III), Dentro de Alphageminivirinae, el género Monogeminivirus (subgrupo I), que presenta transmisión por saltahojas, infecta monocotiledóneas y como miembro tipo: Maize Streak Virus. Los Betageminivirinae contienen tres géneros:Intergeminivirus, Archeogeminivirus y Neogeminivirus. Dentro del género Intergeminivirus están los transmitido por el saltahojas, infectan dicotiledóneas y como miembro tipo: Beet Curly Top Virus (BCTV). Los del género Archeogeminivirus están ubicados en el viejo mundo, transmitidos por la mosca blanca, infectan dicotiledóneas, su genoma puede ser monopartita o bipartita y su miembro tipo: African cassava mosaic virus, mientras que el género Neogeminiviru se ubican en el nuevo mundo, transmitidos por la mosca blanca, infectan dicotiledóneas. Hasta la fecha su genoma es bipartita y su miembro tipo: Bean Golden Mosaic Virus.
La segunda propuesta se basa en el análisis de la secuencia de ADN, esta es similar a la de Padidam y colaboradores (1995), pero con un esquema más simplificado organizándose en tres géneros. Monogeminivirus (subgrupos): hospedantes monocotiledóneos, y saltahojas como vector. Bigeminivirus (subgrupo III): hospedantes dicotiledóneos, mosca blanca como vector y genoma monopartita o bipartita e Intergeminivirus (subgrupo II): hospedantes dicotiledóneos y saltahojas como vector.
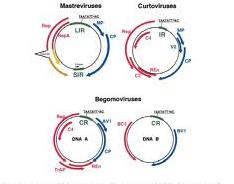
En cuanto al sistema de clasificación hay pocos desacuerdos con respecto a la relación filogenética y la organización de los virus dentro de la familia Geminiviridae. El establecimiento de subtaxas adicionales dentro de la familia es prematuro, por no existir criterios específicos para distinguir cepas y especies. Se utiliza la agrupación en tres géneros dentro de la familia Geminiviridae, utilizando virus tipos para diferenciarlos y para la nomenclatura de los mismos. Mastrevirus (antes subgrupo I), miembro tipo Maize streak virus (MSV), genoma monopartita, vector saltahojas e infectando monocotiledóneas.
Cortovirus (antes subgrupo II), miembro tipo Beet curly top virus (BCTV), genoma monopartita, vector saltahojas e infectando dicotiledóneas y Begomovirus (antes subgrupo III), miembro tipo Bean golden mosaic virus (BGMV), genoma monopartita o bipartita, vector mosca blanca e infectando dicotiledóneas. Más recientemente, siguiendo estos mismos parámetros para la agrupación, se ha definido un cuarto género quedando la familia dividida en: Mastrevirus, Curtovirus, Topocuvirus y Begomovirus .
El género Mastrevirus incluye, hasta la fecha, 41 virus diferentes, de los cuales 24 corresponden a diferentes aislados, cepas u otra subdivisión de MSV. En el género Curtovirus, hay 8 virus diferentes, 5 de ellos relacionados con el BCTV.
El grupo más numeroso corresponde al género Begomovirus, con 122 miembros definidos, 18 de ellos asociados al Tomato yellow virus y 14 al Tomato leaf curl virus .
Sintomatología
Los síntomas ocasionados por geminivirus se pueden confundir con los causados por deficiencias nutricionales y los inducidos por otras familias de virus, principalmente los Tobamovirus y Potyvirus, lo cual dificulta su diagnóstico mediante técnicas convencionales. Estos síntomas varían dependiendo del hospedante, de la etapa en que se adquiere la infección, del geminivirus que se trate y de las condiciones ambientales.
De forma general los síntomas son: enrollamiento foliar, mosaico amarillo brillante, moteado clorótico, clorosis foliar marginal, epinastias, deformaciones foliares como abultamientos y ampollas, reducción del área foliar, enanismo, abscisión floral, amoratamiento foliar, reducción del tamaño de los frutos, entre otros.
En el cultivo del tabaco, dentro de los principales síntomas se encuentra el encrespamiento foliar, las hojas se enroscan y se deforman, formándose ampollas. Estas se engrosan y pueden presentar enaciones de las venas. En los demás miembros de la familia Solanáceae se aprecian síntomas de encrespamiento foliar, amarillamiento y enchinamiento.
Rango hospedante y transmisión
Los principales cultivos agrícolas atacados por geminivirus son: maíz, caña de azúcar, tabaco, remolacha, tomate, fríjol, yuca, algodón, melón, chile, papa, calabaza, sandía, papaya, camote y soya.
En algunos casos el rango hospedante es muy amplio y en otros se restringe a la familia botánica a la cual atacan. En el caso del tabaco encontramos un rango de hospedantes estrecho.
La transmisión para Mastrevirus y Curtovirus es mediante saltahojas y para Begomovirus es por la mosca blanca B. tabaci. Es de tipo circulativo, no propagativo. En la transmisión el insecto adquiere el virus, pasa vía pared del tracto digestivo hasta la hemolinfa y luego a las secreciones salivares y cuando el insecto se alimenta de una planta huésped es inoculado en la misma.
En el caso de los geminivirus transmitidos por mosca blanca en los últimos tiempos han cobrado gran importancia ya que se han logrado adaptar a diferentes medios, estos se sitúan a través de todo el mundo, sobre todo en regiones tropicales y subtropicales, ocasionando problemas en los cultivos tanto desde el punto de vista de vectores altamente eficientes en la transmisión de virus, como por daños directos en los campos actuando como plaga. La causa de las actuales epifitias, se debe a la presencia de un nuevo biotipo o especie de B. Tabaci, que es capaz de desplazar a las poblaciones originales y establecerse en nuevas regiones. Este biotipo presenta alta voracidad para alimentarse, altas tasas de reproducción y un amplio rango de hospedantes donde es muy polífago, además desarrolla rápidamente resistencia a insecticidas, lo que favorece su prevalencia por encima de otros biotipos y dificulta su control.
Efectos citopáticos y localización en los hospedantes
Las infecciones por geminivirus tienen como resultado alteraciones en las células y los organelos celulares, y la aparición de estructuras asociadas al virus en la planta enferma. Los geminivirus mayormente están confinados al floema, los cambios más notables ocurren en el núcleo de la célula infectada.
La patología más común son cuerpos de inclusiones nucleares, estos agregados contienen partículas semejantes a virus y tienen aspectos de cuentas de collar y círculos fibrilares.
Estas estructuras se presentan en el núcleo, en el floema, en el parénquima empalizado y esponjoso, en las células acompañantes y en los elementos cribosos en diferenciación y maduros. El sitio primario de cambios patológicos son los núcleos celulares, que contienen nucleolos mucho más alargados y segregantes. Los cuerpos de inclusión se observan en el citoplasma de las células infectadas rodeando el retículo endoplasmático, el complejo de Golgi y los ribosomas.Se observan vesículas citoplasmáticas, necrosis y degeneración del parénquima vascular y los elementos cribosos, indicando daños extensos en el sistema vascular que se expresan en síntomas de enanismo y reducción del crecimiento de las plantas infectadas.
Distribución y daños
En algunas zonas donde se realiza el monocultivo, el estar aisladas geográficamente y la baja presencia de hospedantes ocasionan epifítias con un solo geminivirus como agente causal. Un ejemplo de ello lo constituye Israel con el Tomato yellow leaf curl virus (TYLCV) donde las pérdidas por este virus son alarmantes. En las zonas tropicales y subtropicales, del área de Mesoamérica y el Caribe, generalmente se presentan infecciones por más de un geminivirus, en estas infecciones mixtas, la interacción puede causar cambios en los síntomas, afectando en mayor o menor grado cada geminivirus implicado. Algunos Begomovirus han cobrado importancia debido al insecto vector, se han reportado grandes epifitias en Solanáceas y en otros cultivos de interés con pérdidas en campos de un 80-90 %.
Muchos de ellos afectan otras solanáceas además del tomate, que son de gran importancia y han llegado a ser limitantes serias en la producción de muchas zonas.
Organización genómica
Los genomas geminivirales están conformados por una (virus monopartitos) o dos (virus bipartitos) moléculas de DNA circular de cadena sencilla de 2.5 a 3.0 Kb de tamaño cada una, sólo durante la replicación se convierten en moléculas de doble cadena. (Hanley-Bowdoin y col., 1999; Lazarowitz, 1992; Palmer y Rybicki, 1998). Cuando los geminivirus son bipartitas, sus componentes genéticos son llamados DNA-A y DNA-B. En el DNA-A se encuentran funciones necesarias para replicación, trascripción y encapsidación, mientras que en el DNA-B son codificadas funciones de movimiento y pueden estar alojados determinantes sintomáticos (Sudarshana y col., 1998; Noueiry y col., 1994).
Cada componente contiene unidades de trascripción divergente, separadas por una región intergénica (RI) de aproximadamente 300 bases. En la región intergénica se localiza una secuencia denominada región común (RC) de aproximadamente 200nt, idéntica para ambos componentes de un mismo geminivirus pero diferentes entre distintos geminivirus. Esta RC contiene un elemento de 30nt con el potencial termodinámico de formar una estructura en horquilla, rica en G-C en el tallo y una secuencia conservada rica en A-T en el asa, la cual contiene el sitio de inicio d la replicación. Además en la RI se encuentra las regiones reguladoras que permiten la expresión coordinada temporal y espacial de los genes. Los geminivirus del genero Mastrevirus tienen genomas monopartitas, son transmitidos por sicadelidos y generalmente infectan monocotiledóneas. Hasta la fecha solamente se han reportado dos Mastrevirus que infectan dicotiledóneas, uno en Australia y otro en Sudáfrica.
Los Begomovirus, en su mayoría presenta genoma bipartitas, con unos pocos ejemplos monopartitas, todos infectan dicotiledóneas y son transmitidos por la mosca blanca. Los Curtovirus, se encuentran como una especie de paso intermedio entre los otros dos géneros, en el sentido en que tienen genomas monopartitas pero infectan dicotiledóneas, siendo transmitidos por sicadelidos y no por mosca blanca. Algunos autores han especulado utilizando bases filogenéticas, que los Curtovirus son el resultado de un posible evento de recombinación entre Mastrevirus arcaicos y virus parecidos a los Begomovirus (Palmer y Rybicki, 19998). El genoma viral se empaqueta dentro de su estructura proteica solamente como una hebra circular de DNA de cadena sencilla que se considera como positiva o de sentido dl virión. Los genes de los geminivirus se nombran por su posición. Los genes en sentido del virión se denotan como una V y los genes en sentido de la cadena complementaria con una C, seguido de un número secuencial.
Para el caso de los Geminivirus bipartitas, el nombre de cada gen es antecedido por la letra A ó B dependiendo del componente genómico en que se encuentre. En el caso de aquéllas proteínas cuya función ya ha sido dilucidada, por ejemplo, se prefiere usar el nombre funcional: Rep, CP, TrAP o Ren. Sin embargo puede ser que resulte precipitado nombrar a las proteínas por su función, en el caso particular de la Rep (AC1) que es una proteína multifuncional (Endonucleas, ligasa, unión a NTPs, posible elicasa, unión a proteínas del huésped, etc:) y no sólo esta involucrada en la replicación, como es el caso de muchas replicasas de virus de RNA (Hanley-Bowdoin, comunicación personal). Pero esta nomenclatura ya ha hecho extensiva.
Proteínas virales
Rep: Es la única proteína viral requerida para la replicación en todos los geminivirus. Muchos geminivirus (Curtovirus y Begomovirus), también codifican para una segunda proteína involucraba en replicación llamada REn, la cual incrementa notablemente la misma. Rep tiene un papel importante en la replicación viral, pero también en la trascripción. Confiere reconocimiento virusespecífico de su origen de replicación correspondiente e indica la replicación del DNA de hebra positiva. Además, reprime su propia expresión a nivel de la transcripción y puede incrementar la trascripción de genes tardíos en algunos geminivirus.
TrAP: El marco de lectura TrAP presente en Begomovirus, codifica par una proteína que transactiva la expresión del gen de la proteína de la cápside y de una de las proteínas de movimiento (BR1). En el caso de bipartitas dicha transactivación parece ser mediada por elementos de secuencia discretos que actúan en cis y que además son específicos para la acción de TrAP.
CP: Es la más abundante de las proteínas virales durante una infección, protege al virión durante su paso por el vector y esta relacionada con la transmisión y especificidad del mismo.
Ren: Potencia significativamente la replicación, aunque no es indispensable para la misma su ausencia reduce en más de 50 veces la tasa replicativa.
BV1 y BC1: Proteínas necesarias para el movimiento del virus dentro de la planta infectada y por tanto para el desarrollo de la infección.
Replicación viral
Una vez que el virión se ha desnudado de la cápsida el primer paso es la síntesis de la cadena complementaria llevando a la producción de ADN de doble cadena el cual es transcripcionalmente activo. Para la síntesis de este ADN complementario se necesita una corta secuencia de ARN la cual es utilizada como primer. La región con homología para esta secuencia se encuentra en la región IGR. En los geminivirus del subgrupo I, los iniciadores para la síntesis de la cadena complementaria son encapsidados en el virión. En estos casos los primers son de aproximadamente 80 nucleotidos de DNA con ribonucleotidos en el extremo 5 terminal (Donson et.al, 1984; Hayes et.al., 1988). En los subgrupos II y III no existen evidencias de que los primers vengan dentro del virion por lo que se piensa que son sintetizados por síntesis de Novo.
En intermediarios de la replicación del ADN en plantas de tabaco infectadas se identificó un indicador de ARN procedente de la síntesis del ADN complementario del African Cassava Mosaic Virus (ACMV) (Saunders, Lucy and Stanley, 1992).
El ADN de doble cadena formado, sirve entonces como molde para la transcripción de los genes de la cadena complementaria, así como para la replicación en círculo rodante. A partir del proceso transcriptivo de esta molécula se producen las proteínas AC1 y AC3 requeridas para iniciar la replicación (Xiong, 1995).
Para la síntesis de la cadena del virión se produce un corte en la molécula de ADN circular en el origen de replicación, esta función es llevada a cabo por la proteína AC1.
En base con la homología del sitio de clivage en el fago X174 se ha sugerido que el sitio de corte se encuentra dentro del nonanucleotido conservado en todos los geminivirus. Lauf y colaboradores (1995) demostró que la proteína AC1 del Tomato yellow leaf curl virus inicia la síntesis de la cadena viral introduciendo un corte en la cadena positiva dentro del nonanucleotido TAATATT/AC. Después del corte la proteína AC1 permanece unida al extremo 5’ de la cadena clivada. El extremo 3’ sirve entonces como iniciador para la síntesis de ADN, desplazando la cadena original. La enzima encargada de esta reacción continuamente gira alrededor del ADN molde siguiendo por tanto el modelo del circulo rodante. Cuando una unidad de la cadena ha sido copiada la misma es cortada y ligada para formar una molécula de ADN circular de simple cadena, la cual puede srvir entonces como molde para otro ciclo de replicación o puede ser encapsidada dentro de los viriones. Si una cadena de una unidad de longitud no es cortada entonces se forman concatámeros (Xiong, 1995).
Estudio y diagnóstico
Actualmente se reportan dos nuevos Begomovirus afectando al tabaco aislados en Zimbabwe y México (Paximadis y col. 1999). La detección de los geminivirus es importante para el manejo de las enfermedades que ocasionan, y puede hacerse detectando la proteína de su cápside o su genoma.
En el área de Mesoamérica y el Caribe el diagnóstico es más complejo, porque a menudo coexisten varios geminivirus en una misma planta. Estas mezclas son dinámicas y presentan una variación temporal y espacial, lo que dificulta el diagnóstico, porque la sintomatología es muy variable. En una temporada en un período de tiempo podrían aparecer dos geminivirus diferentes y posteriormente aparecer otros diferentes a los anteriores o una mezcla de ellos con otros nuevos al mismo tiempo; entonces podría presentarse que cultivares tolerantes a uno de estos virus se infecten completamente con este por la acción de los otros. Es decir, que la presencia de un virus puede hacer que otro infecte cultivares que por si solo no afectaría (Hilje, 1996).
El estudio, caracterización y diagnóstico de los geminivirus se hace difícil en general, se utilizan plantas indicadoras, pero no se conocen hospedantes que muestren síntomas de hipersensibilidad, sólo aquellos susceptibles y otro inconveniente es que en su mayoría no se transmite mecánicamente.
Se diagnóstica por microscopía óptica detectando las inclusiones celulares inducidas por el virus en las células de la planta, estas técnicas dan un diagnóstico poco preciso y general sin tener en cuenta los subgrupos y estirpes del virus. También existe el diagnóstico mediante microscopía electrónica, pero tiene la desventaja que en la preparación se observan muchas cosas, pues no esta purificada, y es difícil reconocer el virus.
El estudio de las interacciones entre geminivirus debido a su situación justifica el desarrollo de métodos de detección específicos. Técnicas como el ELISA han sido muy utilizadas para el diagnóstico de virus, debido a su alta sensibilidad y su bajo costo. Pero esta técnica en geminivirus no es factible porque la concentración de virus en la planta infectada es muy baja, lo que exige manipular gran cantidad de tejido infectado y altos volúmenes de soluciones, lo que disminuye la eficiencia de la purificación. Además, las partículas tienen baja capacidad antigénica, lo que hace que no provoquen una reacción inmunológica fuerte en el animal inoculado obteniéndose bajos niveles
de anticuerpos que no se pueden utilizar para la técnica ELISA (Hilje, 1996).
Aunque existen intentos para la obtención de anticuerpos contra geminivirus con resultados como es el caso del 3F7.
Las técnicas basadas en al manipulación de los ácidos nucleicos son claves para la detección de geminivirus como son: detección visual del ADN viral, hibridación molecular y reacción en cadena de la polimerasa (PCR) además de la detección se necesita una herramienta adicional para detectar geminivirus en general y otra para geminivirus específicos (Hilje, 1996).
Para la extracción del ADN existen diferentes metodologías con requerimientos distintos. Por ejemplo el RFLP es una técnica que se basa en digestión con enzimas de restricción, por lo que se necesitan de cantidades de ADN en el orden de los microgramos. Además es bien conocido de que la mayoría de las endonucleasas son inhibidas por diversos contaminantes por lo que se debe garantizar que el ADN tenga una pureza lo más elevada posible. En los métodos que se basan en PCR y Southern blot las cantidades de ADN a emplear son mínimas y no necesariamente puras, lo que conlleva a que se puedan emplear métodos más rápidos y sencillos.
Como herramientas moleculares más usadas para la detección e identificación de geminivirus transmitidos por mosca blanca, estan las técnicas de PCR (Bird et al., 1995; Brow and Bird, 1992; Howarth and Vandermark, 1989; Paximadis and Rey,1997; Sambrook, Fritsch and Maniatis, 1989) hibridación de ácidos nucleicos (Abouzid, Hiebert, and Stranderg, 1992; Berrie et al., 1998; Hong and Harrison, 1995; Osaki and Inouye, 1981; Padidam, Beachy and Fauquet, 1995; Swofford, 1993), clonación molecular y secuenciación del ADN. Estas últimas la clonación y secuenciación ayudan a la identificación de virus relacionados y de nuevas especies (Arguello- Astorga et al., 1994; Doyle and Doyle, 1987; Orozco et al., 1998; Rybicki, 1994; Torres-Pacheco et al., 1996). Varios programas computacionales seon usados para el análisis de las secuencias, la relación entre dendogramas basados en la secuencia de la CP ayuda a la identificación y al conocimiento del origen de los geminivirus (Macintosh, Robinson and Harrison, 1992; Roberts, Robinson and Harrison, 1984; Rybicki, 1994). Se ha demostrado que porcientos de identidad mayores al 90% en al secuencia de nt de la CP se consideran cepas de la misma especie viral (Macintosh, Robinson and Harrison, 1992).
También los iterones dentro de la región común son un aspecto importante en la identificación, los virus que muestran iterones conservados son virus relacionados entre sí, se encuentran en en la misma rama o próximos en el árbol filogenético (Arguello- Astorga et al., 1994; Arguello- Astorga, Herra and Rivera-Bustamante, 1994; Orozco et al., 1998).
Evidencias recientes indican la existencia en el Viejo Mundo de geminivirus distintos a el Tobacco leaf curl virus TbLCV, asociados a enfermedades de encrespamiento foliar en el cultivo del tabaco (Paximadis y col., 1999), el agente causal de estas enfermedades en tabaco es diverso e iduce síntomas confusos (Rochester et al., 1994; Rojas et al., 1993).
Importancia económica y control
La industria del tabaco constituye un importante segmento de las actividades de negocio en el mundo entero. Por la amplia, insistente y cada vez mayor demanda, que no se afecta fácilmente por los cambios de precio de la hoja, ni por el ingreso del consumidor, a parte que el crecimiento de la población mundial y el número de fumadores, le aportan un mercado en continuo incremento.
Los daños causados por geminivirus inciden considerablemente en la economía pues las hojas de las plantas infectadas generalmente son pequeñas, deformadas y no cosechables.
Además las plantas infectadas son más susceptibles a otros patógenos, particularmente Cercospora.
Su control fundamental es sobre el vector B. tabaci (Genn.). La destrucción de toda la materia viva al termino de cada cosecha. Sembrar en la época óptima según la zona, evitar colindancia con campos más viejos infectados, la eliminación de malezas hospedantes en los bordes de los campos y guardarraya, utilización de transplantes sanos, y la selección negativa de las plantas enfermas en el campo.
Referencias
- Abouzid, A. M. itaHiebert, E. Stranderg, J. O. 1992. Cloning, identification and partial sequencing of the genomic components of a geminivirus infecting the Brassicaceae. Phytopathology 82: 1070.
- Akehurst, B. C. 1973. El tabaco. Ciencia y Técnica. Instituto cubano del libro, La Habana. 682p.
- Altschul, Stephen F., Thomas L. Madden, Alejandro A. Schäffer, Jinghui Zhang, Zheng Zhang, Webb Miller, and David J. Lipman. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25: 3389-3402.
- Arguello- Astorga, G. R. Guevara-Gónzalez, R. G. Herrera-Estrella, L. R. Rivera-Bustamante, R. F. 1994. Geminivirus replication origings have a group-specific organization of iterative elements: a model for replication. Virology 203: 90-100.
- Arguello- Astorga, G. R. Herra L. And Rivera-Bustamante, R. F. 1994. Experimental and theorical definition of geminivirus origin of replication. Plant Mol Biol 26: 553-556.
- Argüello-Astorga, R. A. and R. Ruiz-Medrano. 2001. An iteron-related domain is associated to Motif 1 in the replication proteins of geminiviruses: identitication of potential interacting amino acid-base pairs by a comparative approach. Archives or Virology.
- Ascencio-Ibáñez J. T. Monsalve-Fonnegra, Z. Pruna-Camacho, M. Díaz-Plaza, R. Rivera-Bustamante, R. F. 1999a. Los Geminivirus. Revista Mexicana de Fitopatologia 17 (2): 113-127.
- Ascencio-Ibañez, J. T. Arguello-Astorga, G. R. Mendez-Lozano, J. And Rivera-Bustamante, R. F. 2002. First Report of Rhynchosia golden mosaic virus (RhGMV) infecting Tobacco in Chiapas, México. Plant Disease 86 (6): 692.
- Ascencio-Ibáñez, J. T. Díaz-Plaza, R. Méndez-Lozano, J. Monsalve-Fonnegra, Z. I.
- Argüello-Astorga, R. A. and Rivera-Bustamante. 1999b. First report of Tomato Yellow Leaf Curl Geminivirus in Yucatán, México. Plant Disease 83: 1178.
- Bedford, I. D. Briddon, R. W. Brow, J. K. Rosell, R. C. Markham, P. G., 1994. Geminivirus transmition and biological characterization of Bemisia tabaci (Gennadius) biotypes from different geographyc regions. Ann. Appl. Biol. 125: 311-325.
- Berrie, L. C. Palmer, K. E. Ribicki, E. P. Rey, M. E. C. 1998. Molecular characterization of a distinc South African cassava infecting geminivirus. Arch Virol 143: 1-8.
- Bird, J. Sánchez, J. Rodríguez, R. L. Julia, P. J. 1995. Rugaceus viruses in Puerto Rico. In: Maramarosch K. Bird J (eds) Tropical desease of legumes. Academic Press. New York. pp 2-25.
- Briddon, R. W. Bedford, I. D. Tsai, J. H. Markham, P. G. 1996. Analysis of the nucleotide sequence of the treehopper-transmitted geminivirus, tomato pseudomcurly top virus, suggest a recombinat origin. Virology 219 : 387-394.
- Brow, J. K. Bird, J. 1992. Whitefly-transmitted geminiviruses and associated disorders in the Americas and the Caribbean Basin. Planst Dis 76: 220-225.
- Brown, J. K. 1996. Chapter 5 in: Molecular Biology and Epidemiology of Subgroup III, Geminiviridae. Plant-Microbe Interactions Review Series, G. Stacey and N. Keen, eds (Chapman and Hall) pp 125-195.
- Brown, J. K. 2000. Molecular markers for the identification and global tracking of whitefly vector-Begomovirus complexes. Virus Research 71:233-260.
- Christie, R. J. Ko, N. J. Falk, J. B. W. Hiebert, H. Lastra, R. Bird, J. and Kim. K. S. 1986. Light microscopy of geminivirus-induced nuclear inclusion bodies. Phytopathology 76: 124-126.
- Costa, A. S. 1976. Whitefly-transmitted geminivirus. Plant Diseases. Annual Review of Phytopathology 14:429.
- Crespi, S., Accoto, G. P., Caciagli, P., and Gronenborn. B. 1991. Use of digoxigeninlabelled probes for detection and host-range studies of tomato yllow leaf curl geminivirus. Res. Virol. 142:283-288.
- David Shew, H. and Lucas, G. B. 1996. Compendium of tobacco disease. APS PRESS. The American Phytopathological Society. http://www.shopapspress.org/41175.html.
- Dellaporta, S.L., Wood, J. and Hicks, J.B. 1983. A plant DNA minipreparation: Version II. Plant Mol.Biol. Rep.1:19-21.
- Dhar, A. K. and Singh, R. P. 1995. Geminiviruses, in: Pathogenesis and Host Specifity in Plant Disease, Vol. III, Viruses and Viroids. Singh y Kolimoto, Eds. Pergamon Press, U. K. 289-307.
- Donson, J., Morris-Krsinich, B. A. M., Mullineaux, P. M., Boulton, M., and Davies, J. W. 1984. A putative primer for second strand DNA synthesis of maize streak virus is virion associated. EMBO. 3, 3069-3073.
- Doyle, J. J. Doyle, J. L. 1987. A rapid isolation procedure for small quantities of fresh leaf tissue. Phytochem Bull 19: 11-15.
- Fauquet C. M., D. P. Maxwell, B. Gronenborn, and J. Stanley. 2000. Revised proposal for naming geminiviruses. Arch Virol 145(48): 1743-1761
- Garzón- Tiznado, J. A. Torres-Pacheco, I. Ascencio-Ibáñez, J. T. Herrera-Estrella, L. and Rivera-Bustamante. 1993. Inoculation of peppers with infectious clones of a new Geminivirus by a biobalistic procedure. Phytopathology 83: 514-521.
- Gilberson (1991) Hanley-Bowdoin, L. Settlage, S. B. Orozco, B. M. Nagar, S. Robertson, D. 1999.
- Geminiviruses: Model for plant DNA replication, Transcription and cell cycle regulation. Crit. Rev. Plant Sci. 18: 71-106.
- Harrison, B. D. 1985. Advances in geminivirus Research. Annual Review of Phytopathology 23:55-82.
- Hartitz, D. M; G. Sunter and D. M. Bisaro. 1999. The Tomato Golden Mosaic Virus Transactivator (TrAP) is a Single-Stranded DNA and Zinc-Binding
- Phosphoprotein with an Acidic Activation Domain. Virology. 263, 1-14.
- Hayes,R.J., Petty,I.T.D., Coutts,R.H.A., and Buck,K.W. 1988. Gene amplification and expression in plants by a replicating geminivirus vector. Nature 334, 179-182.
- Heyraud-Nitschke, F. Scumacher, S. Laufs, J. Schaefer, S. Schell, J. and Gronenborn, B. 1995. Determination of the origin cleavage and joining domain
- of geminivirus rep proteins. Nucleic Acids Research 23: 910-916.
- Hilje, Luko. 1996. Metodología para el estudio y manejo de moscas blancas y Geminivirus. Ed.- Turrialba, Costa Rica: CATIE. Unidad de Fitoprotección. 150p.
- Hong, Y. G. Harrison, B. D. 1995. Nucleotide sequence from tomato leaf curl viruses from different countries: evidences for three geographically separate brances in
- evolution of he coat protein of whitefly-transmitted geminiviruses. J Gen Virol 76: 2043-2049.
- Howarth, A. J. Vandermark, G. J. 1989. Phylogeny of geminiviruses. J. Gen Virol 70: 2717-2727.
- ICTV Database. 2002a. Begomovirus. http://life.anu.edu.au/viruses/ICTVdB/29000000. Htm.
- ICTV Database. 2002b. Geminiviridae. http://life.anu.edu.au/viruses/ICTVdB/29000000. Htm.
- Jeske, H. and G. Schuchalter-Eicke. 1984. The Abutilon mosaic virus (AbMV) in diferent leaf tisúes of several host species of Malvaceae. Phytopathol. Z. 109:353-362.
- Kim, k. and E. M. Flores. 1979. Nuclear changes associated whit euphorbia mosaic virus transmitted by the whitefly. Phytopathology. 69: 980-984.
- Kim, K. S. y R. W. Fulton. 1984. Ultrastructure of Datura stramonium infected with a Euphorbia virus suggestive of a whitefly-transmitted geminivirus. Phytopathology
- 74: 236-241.
- Kim, K. S., T. L. Shock, y R. M.Goodman. 1978. Infection of Phaseolus vulgaris by bean golden mosaic virus: Ultrastructural aspects. Virology. 89: 22-33.
- Krake, L. R. Rezaian, M. A. and Dry, I. B. 1998. Expression of the Tomato Leaf Curl Geminivirus C4 Gene Produces Viruslike Symptoms in Transgenic Plants.
- Laufs, J. Traut, W. Heyraud, F. Matzeit, V. Rogers, S. G. Schell, J. Gronenborn, B. 1995. In vitro cleavage and joining at the viral origin of replication by replication
- initiator protein of tomato yellow leaf curl virus. Proc Natl Acad Sci USA 92: 3879-3883.
- Lazarowitz, S. G. 1992. Geminiviruses: genome structure and gene-function. Critical reviews in plant sciences 11, 327-349.
- Liu, L. van Tonder, T. Pietersen, G. Davies, J. W. Stanley, J. 1997. Molecular characterisation of a subgroup I geminivirus from a legume in South Africa. J Gen Virol 78: 2113-2117.
- Lucas, G. B. 1965. Enfermedades del tabaco. (2. ed.) Capitulo 39. Enroscamiento de la hoja. La Habana. Ed. Rev. Inst. del Libro.
- Macintosh, S., Robinson, D. J., and Harrison, B. D. 1992. Detection of three whiteflytransmitted geminiviruses occurring in Europe by tests with heterologous monoclonal antibodies. Ann. Appl. Biol. 121:297-303.
- Markham, P. G. Bedfor, I. D. Liu, S. and Pinner, M. S. 1994. The Transmission of Geminiviruses by Bemisia tabaci. Pesticide Science 42: 123-128.
- Morales, F. J. and Anderson, P. K. 2001. The emergence and dissemination of whitefly-transmitted geminiviruses in Latin America. Arch Virol 146: 415-441.
- Morales, F. G. 1998. The Emergence of Whitefly-Transmitted Geminiviruses as important pathogen of cultivated plants in Latin America. Abstract. International Workshop on Bemisia and Geminivirus. San Juan, Puerto Rico. L-81.
- Muniyappa, V. 1980. Whitefly. In: Harris, K. F. Maramorosch, K. (Eds.), Vectors of Plant Pathogens. Academic Press, New York, pp. 39-85.
- Muniyappa, V., Swanson, M. M., Duncan, G. H., and Harrison, B. D. 1991. Particle purification, properties and epitope variability of Indian tomato leaf curl geminivirus. Ann. Appl. Biol. 118:595-604.
- Murphy, F. A. Fauquet, C. M. Bishop, D. H. L. Ghabrial, S. A. Jarvis, A. W. Martelli, G. P. Mayo, M. A. and Summers, M. D. (Eds.). 1995. Virus Taxonomy. Classification and Nomenclature of Viruses. Sixth Report of the International Committee on Taxonomy of Viruses. Springer, Wien New York (Archives of Virology, Suppl. 10).
- Noueiry, A. O. Lucas, W. J. Gilbertson, R. L. 1994. Two proteins of a plant DNA virus coordinate nuclear and plasmodesmatal transport. Cell 76. 925-932.
- Orozco, B. M. Glandfelter, H. J. Settlage, S. B. Eagle, P. A. Gentry, R. N. Hanley-Bowdoin. L. 1998. Multiple Cis elements contribute to geminiviru origin function.Virology 242: 346-356.
- Osaki, T. Inouye, T. 1981. Tobacco leaf curl virus. CMI/AAB Descriptions of Plant Viruses. No. 232.
- Padidam M, Beachy RN and Fauquet. 1995. Classification ad identification of geminiviruses using sequence comparisons. J Gen. Virol. 76:249-263.
- Padidam, M. Maxwell, D. P. and Fauquet, C. M. 1997. A proposal for naming geminiviruses. Virology Division News 142 (12): 2553-2562.
- Page, R. D. M. 1998. TREEVIEW: An application to display phylogenetic trees on personal computers. Computer Applications in the Biosciences 12: 357-358.
- Palmer, K. E. and Rybicki, E. P. 1998. The Molecular Biology of Mastrevirus. Advances in Virus Research 50: 183-233.
- Paximadis, M. Idris, A. M. Torres-Jerez, I. Villarreal, I. Rey, M. E. C. Brown, J. K. 1999. Characterization of geminivirus of tobacco in the Old and New World. Arch. Virol. 144, 703-717.
- Paximadis, M. Rey, M. E. C. 1997. Aetiology of tobacco leaf curl in Southern Africa. Ann Appl Biol 131: 449-457.
- Polston, J. E. Anderson, P. K. 1997. The emergence of whitefly-transmitted geminiviruses in tomato in the Western hemisphere. Plant Dis. 81: 1358-1369.
- Quintero, S. y Santiesteban, J. 1979. Virus del encrespemiento foliar de tabaco (tobacco leaf curl virus). Agrotecnia de Cuba. 11(1): 61-74.
- Ramírez, P. y Bustamante, R.. 1996. Metodología para el estudio del complejo mosca-blanca-geminivirus. Capitulo 4: Identificación de geminivirus.
- Roberts, I. M. Robinson, D. J. and Harrison, B. D. 1984. Serological relationships and genome homologies among geminiviruses. Ann. Appl. Biol. 105: 483-493.
- Rochester, D. F. Fauquet, C. M. DePaulo, J. J. and Beachy, R. N. 1994. Complete nucleotide sequence of the geminivirus, tomato yellow leaf curl virus (Thailand isolate). J. Gen. Virol. 75: 477-485.
- Rojas, M. R., Gilbertson, R. L., Russell, D. R., Maxwell, D. P.1993. Use of degenerate primers in the �olimerasa chain reaction to detect whitefly transmitted geminiviruses. Plant Disease 77:340-347.
- Rybicki, E. P. 1994. A phylogenetic and evolutionary justification for three genera of Geminiviridae. Arch Virol 139: 49-77.
- Sambrook, J. Fritsch, E. F. Maniatis, T. 1989. Molecular cloning-a laboratory manual, Vols I, II, III, 2nd ed. Cold Spring Harbour Laboratory Press, New York.
- Sanderfoot, A. A. and Lazarowitz, S. G. 1996. Getting it all together in plant virus movement: cooperative interactions between bipartite geminivirus movement proteins. Trends Cell Biol. 6: 353-358.
- Sanger, F. Nicklen, S. And Coulson, A. R. 1977. DNA sequencing with chainterminating inhibitors. Proc. Natl. Acad. Sci. U. S. A. 74: 5463-5467.
- Saunders, K., Lucy, A., and Stanley, J. 1992. RNA-primed complementary-sense DNA synthesis of the geminivirus African cassava mosaic virus. Nucl. Acids.
- Sudarshana, M.R. Wang, H. L. Lucas, W. J. And Gilbertson, R. L. 1998. Dynamics of Bean Dwarf Mosaic Geminivirus cell-to-cell and long-distance movement in
- Phaseolus vulgaris revealed, using the green fluorescent protein. Molecular Plant-Microbe Interactions 11 (4): 277-291.
- Swanson, M. M., and Harrison, B. D. 1993. Serological relationship and epitope profiles of an isolate of okra leaf curl geminivirus from Africa and the Middle East. Biochimie 75:707-711.
- Swanson, M. M., Brown, J. K., Poulos, B. T., and Harrison, B. D. 1992. Genome affinities and epitope profiles of whitefly-transmitted geminiviruses from the Americas. Ann. Appl. Biol. 121:285-296.
- Swofford, D. L. 1993. PAUP: Phylogenetic analysis using parsimony. Illinois Natural History Survey, University of Illinois, Champaign.
- Thompson, J.D., Higgins, D.G. and Gibson, T.J. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, positions-specific gap penalties and weight matrix choice. Nucleic Acids Research, 22:4673-4680.
- Torres-Pacheco, I., J. A. Garzón-Tiznado, J. K. Brown, A. Becerra-Flora and F. And Rivera-Bustamante. 1996. Detecction and distribution of geminiviruses in México and the Southern United States. Phytopathology. Vol 86. 11: 1186-1192.
- van Regenmortel, M. H. V. Fauquet, C. M. Bishop, D. H. L. Carstens, E. Estes, M. K. Lemon, S. Maniloff, J. Mayo, M. A. McGeoch, D. Pringle, C. R. and Wickner, R.
- B. (eds). 2000. Virus taxonomy: seventh report of the International Committee on Taxonomy of Viruses. Academic Press, London, San Diego.
- Wyatt, S. D. and Brown, J. K. 1996. Detection of subgroup II geminiviruses isolate in leaf extracts by degenerate primers and polymerase chain reaction. Phytopathology 86: 1288-1293.
- Xiong, Z. 1995. Single stranded DNA viruses: Geminiviruses. http://ag.arizona.edu/%zxion/plp611/lect17.html.
Fuentes
- Geminivirus(PDF)
- Los geminivirus(PDF)
- Gemiivirus de Tomate(PDF)
- La presencia del geminivirus(PDF)
- La infección del geminivirus
- Muestra del geminivirus
- EL geminivirus azota Costa Rica(PDF)
- Geminivirus
- http://dialnet.unirioja.es/servlet/articulo?codigo=300558
- http://www.caripestnetwork.org/vtt/docs/datasheets/viruses/gemini_virus_complex.pdf (PDF)
- http://orton.catie.ac.cr/repdoc/A2012E/A2012E.PDF (PDF)
- http://dnareplication.cshl.edu/content/free/chapters/30_bisaro.pdf (PDF)