Microsatélite
| ||||||
Microsatélite. Pequeñas secuencias o fragmentos del ADN (ácido desoxirribonucleico), repetidas n veces una a continuación de la otra o en tándem, tienen apenas entre 2-10 pares de bases (pb) o más frecuentemente, de 2-6 pb. Son altamente polimórficos, muy abundantes y están dispersos por el genoma de eucariontes y en algunos procariontes.
Es común que se les identifique como secuencias simples repetidas (del inglés, simple sequence repeats-SSR) o STR (short tandem repeats). Debido a sus características y múltiples ventajas, se han convertido en una técnica para la detección de marcadores moleculares con diversas aplicaciones en el campo de la genética de plantas, humana y animal; de ella se conocen diferentes variantes o modificaciones.
Sumario
Descubrimiento
Si bien los microsatélites fueron inicialmente descritos en humanos en 1989 (Litt y Lutty, 1989), su potencial como marcadores útiles para los estudios en plantas fue rápidamente reconocido, lo que explica su amplio desarrollo durante los últimos años en muchas especies, convirtiéndose en una técnica estándar para la evaluación genética (molecular) de cualquier población.
Sin embargo, los marcadores de este tipo generan un gran volumen de información debido al número y frecuencia de los alelos que detectan y tienen una alta repetitividad. Esto los convierte en poderosos marcadores muy útiles en plantas, animales y humanos; inicialmente descritos en humanos, rápidamente fue reconocido su potencial en muchas especies, especialmente en plantas, con un desarrollo vertiginoso en los últimos años.
Generalidades
En el genoma existe un ADN ubicuo y abundante denominado "microsatélite" que consiste en repeticiones en tándem (repetidas y adyacentes) de solo unos pocos pares de bases y de un elevado polimorfismo. Los microsatélites o SSR son secuencias simples de ADN con un alto grado de hipervariabilidad, abundantes y bien distribuidas en todo el genoma, que conforman las llamadas familias de ADN satélite repetido; se plantea que ese tipo de ADN puede interactuar con proteínas. Ellos pueden consistir en: dinucleótidos (AC)n, (AG)n o (AT)n (más abundantes en plantas); trinucleótidos (TTG)n, (TCT)n, (TTA)n ó tetranucleótidos (TATG)n, e incluso, pueden llegar a ser penta o hexanucleótidos, donde n es el número de veces que se repite el segmento dentro de un locus, incluso pueden estar en diferentes loci.
Aunque los microsatélites se han usado ampliamente en genética molecular, no se ha identificado bien su función en el genoma, los datos sugieren que ellos desempeñan un rol importante en la estabilización y el mantenimiento de la estructura cromosomal, o quizás en funciones de regulación; una hipótesis bastante aceptada plantea que ellos intervienen en el empaquetado y la condensación del ADN dentro de los cromosomas de eucariotas.
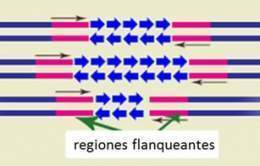
Los microsatélites se utilizan con creciente frecuencia como marcadores genéticos, ya que sus extremos o secuencias flanqueantes son habitualmente bien conservadas y eso constituye la base para el diseño de primers o cebadores, elemento clave para su amplio uso como marcadores moleculares. Generalmente se encuentran en zonas no codificantes del ADN (tienen baja frecuencia en las regiones codificantes), son neutros, codominantes (permiten la distinción de homocigotos y heterocigotos) y poseen una alta tasa de mutación, lo que los hace muy polimórficos. A pesar de su alta tasa de mutación, el hecho de que las regiones flanqueantes estén más conservadas hace que se puedan emplear para la amplificación específica de los alelos de cada locus.
La eficiencia técnica y potencial de los microsatélites los hace preferibles para los análisis genéticos. La razón principal del incremento en el uso de ellos es que proveen la más alta tasa de polimorfismo o PIC (polymorphic information content) en comparación con otras técnicas moleculares, además de su multialelismo, amplia cobertura del genoma y confiabilidad en la diferenciación de genotipos, que los hace atractivos como marcadores genéticos y facilita la integración de los resultados de estudios independientes. Si bien el diseño de SSRs consume tiempo y puede ser costoso, la interpretación de los resultados es sencilla y su reproducibilidad es muy alta, los resultados son transferibles entre laboratorios y es posible su automatización, además de obtenerse por PCR, por lo que se necesitan pequeñas cantidades de ADN.
Clasificación
Además de su definición en función de la cantidad de nucleótidos que se repiten (di-, tri-, tetranuclétidos), también se les puede clasificar en:
a) Microsatélites perfectos o puros: repeticiones en un número mínimo de unidades sin interrupción y sin repeticiones adyacentes (CACACACACACACACACACA) ó (CA)10.
b) Microsatélites compuestos: dos o más repeticiones con un número mínimo de unidades repetidas ininterrumpidas (CACACACACACACACACAGAGAGAGAGAGAGAGAGA) ó (CA)9(GA)9.
c) Microsatélites interrumpidos: repeticiones ininterrumpidas con un número mínimo de repeticiones y separadas por un máximo de cuatro pares de bases (CACACACACACACACACACATTTCACACACACACACACACACACA) ó (CA)10 TTT (CA)11.
Existen varias formas de uso lo que determina la existencia de diferentes variantes de una misma técnica base que amplifica fragmentos genómicos por PCR (Reacción en Cadena de la Polimerasa-PCR) a partir de secuencias microsatélites usadas como cebadores. Todas cumplen tres principios esenciales (Coto y Cornide, 2002):
- Las copias adyacentes de una repetición específica deben estar en sentido inverso dentro de la distancia límite en el genoma para la amplificación.
- Cualquier secuencia repetitiva debe estar ampliamente representada y distribuida en el genoma.
- El polimorfismo que se detecta es el resultado de inserciones/deleciones entre secuencias repetidas conservadas o por variación en el sitio del cebador.
Las variantes más conocidas son:
SAMPL – Selective Amplification of Microsatellite Polymorphic Loci o Amplificación selectiva de loci polimórfico de microsatélites
SSLP – Simple Sequences Length Polymorphism (Polimorfismo de la longitud del microsatélites) o MP-PCR (Microsatellite-Primed PCR, PCR directo con microsatélites).
ISA – Inter-SSR Amplification o Amplificación entre microsatélites
RAMP – Random Amplified Microsatellite Polymorphism o Polimorfismo de microsatélites amplificados al azar.
Antes se habían diseñado SSRs a partir de librerías de ADN genómico (cADN) (Mba et al., 2001) o de ESTs (Raji et al., 2009) pero con el objetivo de perfeccionar la técnica también se han diseñado microsatélites basados en el polimorfismo de la longitud de los fragmentos amplificados (M-AFLP) con aplicaciones interesantes (Acquadro et al., 2005).
Descripción de la técnica molecular de los microsatélites
Un microsatélite esta típicamente conformado por un fragmento repetitivo, dentro del cual se encuentra la secuencia repetida, y dos regiones flanqueantes, las que se encuentran a ambos lados de dicho fragmento repetitivo. Para que un microsatélite sea considerado útil como marcador molecular, toda la variación de la secuencia o polimorfismo debe hallarse dentro de dicho fragmento repetitivo, y al mismo tiempo, las regiones flanqueantes deben estar altamente conservadas al punto de no presentar ninguna variación de secuencia.
A partir de esos supuestos, se diseñan primers o cebadores (también llamados iniciadores) es decir, fragmentos de ADN que tienen la misma secuencia que los extremos de las regiones flanqueantes, para poder amplificar (o producir un alto número de copias) del microsatélite a través de una reacción tipo PCR sobre el ADN de la especie o población en estudio.
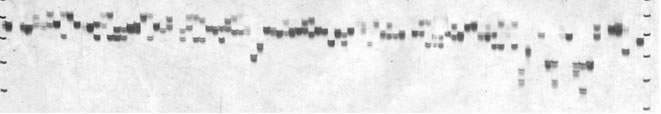
Los fragmentos que se producen son separados de acuerdo a su longitud en pares de base fundamentalmente, a través de una electroforesis en gel de poliacrilamida (PAGE) (Figura 1); ciertos alelos SSR con suficiente diferencia de tamaño pueden ser visualizados mediante un simple gel de agarosa a una concentración óptima (2-3%) y posterior tinción con bromuro de etidio (Figura 2).
También se pueden emplear analizadores de fragmentos más sofisticados como secuenciadores automáticos, basados en electroforesis capilar. El empleo de diferentes flourocromos para el marcaje de los cebadores permite la detección por emisión de fluorescencia, al mismo tiempo, de varios productos de PCR, es decir de varios marcadores. Posteriormente se ha usado la detección por HRM (high resolution melting) en el caso de los SSR y los SNPs (single nucleotide polymorphism o polimorfismo de nucleótido simple). Este método implica la desnaturalización gradual de los amplicones y la detección de pequeños cambios en la fluorescencia emitida gracias al uso de fluoróforos de unión a ADN de doble cadena.
Partiendo de la hipótesis de que en un microsatélite sólo varía el número de repeticiones dentro del motivo repetitivo; fragmentos que tienen el mismo tamaño, tienen la misma secuencia, de manera que todos los fragmentos de un mismo tamaño representarían un alelo; estos productos de la amplificación se visualizan en el gel mediante tinción de plata. Así, cada individuo tendrá un patrón único que permite su discriminación de los demás. La información que genera cada gel se compila y procesa mediante potentes herramientas estadísticas para arribar a conclusiones.
Estos alelos se heredan de manera codominante, es decir, que en cada locus un individuo podría presentar uno o más alelos, dependiendo del número de juegos de cromosomas que posea. Por ejemplo, en una especie diploide con reproducción sexual, hay dos juegos completos de cromosomas y por lo tanto para un locus microsatélite puede presentarse un alelo (si ambos progenitores nos transmitieron alelos de la misma secuencia y tamaño) o dos alelos (si de cada progenitor se heredó un alelo de tamaño diferente). Eso da como consecuencia de que dos individuos puedan ser diferentes entre si.
Principales aplicaciones
Son utilizados como marcadores moleculares en una gran variedad de aplicaciones en el campo de la genética como determinación de relaciones de parentesco y estudios de poblaciones.
Entre las más importantes están los estudios de diversidad genética, en los cuales son los marcadores de elección para la caracterización de germoplasmas vegetales (identificación de duplicados o de progenitores), para analizar variación en poblaciones naturales para el estudio de la estructura y diversidad en poblaciones de diferentes lugares, regiones o países; en estudios dirigidos a cuestiones de biología evolutiva y de la conservación. Han sido útiles para estudios de identificación y en trabajos sobre diversidad genética de razas domésticas o de especies de animales en peligro de extinción (evaluación comparativa de pequeñas poblaciones aisladas).
En general, son herramientas apropiadas en la estimación de relaciones genéticas (distancias genéticas) entre diferentes poblaciones (análisis poblacionales, estudios de diversidad e identificación varietal), en genética forense, pruebas de paternidad (la posibilidad de poder detectar ambos alelos los hace muy útiles para identificaciones individuales en humanos), construcción de mapas genéticos y estudios de asociación.
Otra aplicación, de amplio uso actual es la construcción de mapas de ligamiento más completos y detallados; así como la identificación de genes de interés o QTL (quantitative trait loci). (conocida como cartografía genética). Así, un mapa genético bien saturado de marcadores se convierte en una herramienta muy útil para identificar genes responsables de caracteres de interés. Se trata de buscar asociación entre varios alelos, en cualquiera de los marcadores y en poblaciones que presentan el carácter de interés, para identificar regiones del genoma donde es más probable que se encuentre el gen responsable de ese carácter. Para ello puede conocerse con los SSR la estructura genética de una población y a partir de aquí, utilizar otras técnicas más específicas de mapeo de asociación como las secuencias TAGs expresadas o ESTs (expressed sequence targets), el polimorfismo de nucleótido simple o SNPs (single nucleotide polymorphisms), o los propios SSR con secuencias más específicas. En ese sentido, son múltiples los resultados obtenidos o en ejecución para potenciar el mejoramiento asistido por marcadores en varios cultivos, ya sea directamente con los SSR o a partir de los resultados a los que se ha arribado con ellos, entre ellos la identificación de genes importantes asociados a enfermedades u otras características de interés biológico, ecológico o económico.
En plantas existen interesantes y amplios estudios de diversidad en varios países y en varios cultivos de importancia económica, entre ellos: maíz, maní, trigo, arroz, yuca, forestales, hortalizas y ornamentales. Especialmente importantes son los estudios de diversidad genética desarrollados en la yuca cultivada de Brasil, Cuba, Nigeria, Tanzania, Ghana, Perú y Guatemala, entre otros (disponibles en:
[1]), los cuales proveen de experiencia y herramientas para extender el estudio por países a otras regiones productoras, y así entender y utilizar mejor el potencial genético del cultivo.
Otros aspectos de interés
En la estimación de la estructura poblacional es muy importante tener en cuenta en los análisis el hecho de que los microsatélites presentan una alta tasa de mutación. Dos modelos de mutación básicos existen para los microsatélites: el de alelos infinitos, en el cual cada mutación crea un nuevo alelo a una tasa dada m, se caracteriza por no permitir la homoplasia y porque los alelos idénticos tienen el mismo origen. El modelo extremo de la mutación por pasos (step model mutation, SMM), donde cada mutación crea un nuevo alelo por adición o deleción de una unidad repetida única del microsatélite, con igual probabilidad (m/2) en ambas direcciones, así los alelos de tamaños muy diferentes son menos relacionados que alelos de tamaños similares (Balloux y Lugon-Moulin, 2002).
Otros enlaces
Imagen de microsatélites tomada de [2].
Fuente
- Balloux F y Lugon-Moulin, N. 2002. The estimation of population differentiation with microsatellite markers. Molecular Ecology 11: 155 – 165.
- Beovides, Y. (2008) Análisis de diversidad genética en una colección de trabajo del germoplasma cubano de yuca (Manihot esculenta Crantz). Informe Premio Provincial CITMA, Villa Clara, Cuba.
- Brondani C., R. Brondani, L. Garrido y M.E. Ferreira (2000) Development of microsatellite markers for the genetic analysis of Magnaporthe grisea. Genet. Mol. Biol, 23(4):753-762.
- Coto O. y M.T. Cornide (2002) Principales aplicaciones de los marcadores moleculares. En: Marcadores Moleculares. Nuevos horizontes en la genética y la selección de plantas. La Habana: Ed. Félix Varela. pp. 92-110.
- Ferguson, M; Rabbi, I.; Kim, D.-J.; Gedil, M.; Becerra, L.A. y Okogbenin, E. Molecular markers and their application to cassava breeding: past, present and future. Trop. Plant Biol. 2012, 4: 95-109.
- Okogbenin E, J. Marin, M. Fregene (2006) An SSR based molecular genetic map of cassava. Euphytica 147(3): 433-440.
- Picó M.E. y C. Esteras (2012) Marcadores moleculares basados en PCR: Marcadores SSR o STR (Simple Sequence Repeats or Short Tandem Repeats). Microsatélites. Departamento de Biotecnología, Universidad Politécnica de Valencia (España), 11p.
- Acquadro, A; E. Portis; E Albertini y S Lanteri (2005) M-AFLP- based protocol for microsatellite loci isolation in Cynara cardunculus L. (Asteraceae). Mol. Ecol. Notes., 5:272-274.
- Raji AAJ, JV Anderson, OA Kolade, ChD Ugwu, AGO Dixon e IL Ingelbrecnt (2009) Gene-based microsatellites for cassava (Manihot esculenta Crantz): prevalence, polymorphisms, and cross-taxa utility. BMC Plant Biol., 9: 118.
- Mba R. E. C., P.Stephenson, K.Edwards, S.Melzer, J.Nkumbira, U.Gullberg, K.Apel, M.Gale, J.Tohme, M.Fregene (2001) Simple sequence repeat (SSR) markers survey of the cassava (Manihot esculenta Crantz) genome: towards an SSR-based molecular genetic map of cassava. Theoret. Appl. Genet. 102: 21-31.
- Litt M. y J.A.Lutty (1989) A hypervariable microsatellite revealed by in vitro amplification of dinucleotide repeat within the cardiac muscle actin gene. Am J. Hum Genet. 44: 397-401.||